Visualisasi struktur molekul merupakan hal mendasar bagi biologi molekuler, yang membantu dalam memahami proses biologis yang kompleks. Meskipun kemajuan dalam visualisasi molekuler telah sangat meningkatkan representasi struktur ini, ketidakpastian yang melekat—seperti ketidakakuratan dalam posisi atom atau variabilitas dalam klasifikasi struktur sekunder—mempengaruhi keakuratan visualisasi. Visualisasi yang menyadari ketidakpastian (UAV) muncul sebagai respons terhadap tantangan ini, yang mengintegrasikan ketidakpastian ke dalam representasi visual untuk meningkatkan interpretasi data dan pengambilan keputusan. Meskipun banyak penelitian tentang visualisasi molekuler dan ketidakpastian (UV), masih belum ada survei komprehensif yang membahas persimpangan kedua bidang ini. Makalah ini memberikan tinjauan mutakhir tentang pendekatan UAV untuk struktur biomolekuler. Kami mengusulkan skema klasifikasi yang mengatur metode yang ada berdasarkan jenis molekul yang divisualisasikan, manifestasi ketidakpastian, dan pemetaan ketidakpastian ke representasi visual. Dengan menggunakan kerangka kerja ini, kami mengidentifikasi kesenjangan penelitian dan area untuk eksplorasi masa depan dalam visualisasi struktur biomolekuler yang menyadari ketidakpastian.
1. Pendahuluan
Visualisasi struktur molekuler telah lama menjadi bagian integral dari biologi molekuler struktural dan terus berkembang sebagai alat penting dalam penelitian ilmiah dan industri. Sejak awal bidang ini, visualisasi molekuler telah memungkinkan para peneliti untuk mendapatkan pemahaman intuitif tentang sistem molekuler kompleks yang jika tidak demikian tidak akan dapat diakses melalui data numerik mentah saja. Representasi struktural memberikan wawasan tentang bentuk dan fungsi molekul, yang secara inheren saling terkait. Contoh yang terkenal adalah metafora kunci dan gembok untuk docking molekuler, yang, meskipun disederhanakan, menggambarkan pentingnya bentuk molekuler dalam menentukan interaksi biologis. Dengan mengubah data abstrak menjadi model visual yang dapat ditafsirkan, visualisasi memungkinkan para ilmuwan untuk mengidentifikasi hubungan, mekanisme, dan pola yang penting untuk memajukan biologi molekuler, desain obat, dan bioinformatika.
Levinthal [ Lev66 ] adalah orang pertama yang menggunakan posisi atom yang diketahui, yang diperoleh dari studi difraksi sinar-X awal [ Ken61 , Per64 ], untuk memvisualisasikan gambar struktur 3D protein kecil secara interaktif. Selama bertahun-tahun, visualisasi molekuler telah maju secara signifikan. Bidang ini telah melihat perkembangan beragam teknik visualisasi dan analisis visual, didorong oleh kemampuan komputasi yang berkembang dan meningkatnya kompleksitas data biologis. Kemajuan ini tercermin dalam survei komprehensif seperti yang dilakukan oleh Kozlíková et al. [ KKF*17 ], yang memberikan gambaran rinci tentang keadaan terkini dalam visualisasi struktur molekuler.
Salah satu tantangan inheren dengan visualisasi adalah potensi pengenalan ketidakpastian pada setiap tahap alur visualisasi, dari akuisisi data hingga visualisasi akhir dan interpretasinya oleh pemirsa [ BAOL12 ]. Ada banyak sumber ketidakpastian yang potensial. Misalnya, posisi atom yang diperoleh bisa jadi tidak akurat karena ketidakpastian dalam proses akuisisi data. Contoh penting lainnya adalah variabilitas dalam konformasi molekuler, seperti yang direpresentasikan oleh ensembel. Ensembel yang diperoleh dari teknik seperti spektroskopi resonansi magnetik nuklir (NMR) menggambarkan berbagai kemungkinan struktur daripada konformasi tunggal yang statis. Demikian pula, sifat turunan seperti fluktuasi akar kuadrat rata-rata (RMSF), yang bergantung pada posisi atom selama simulasi dinamika, juga dapat rentan terhadap ketidakpastian. Bentuk ketidakpastian lainnya berasal dari metode komputasi seperti penugasan elemen struktur sekunder: Pengklasifikasi berbeda yang menugaskan elemen struktural ke bagian urutan asam amino menghasilkan hasil yang berbeda. Memahami ketidakpastian ini penting karena secara langsung memengaruhi keandalan dan interpretasi visualisasi molekuler. Kembali ke contoh docking molekuler sebelumnya, ketidakpastian dalam posisi atom dapat sangat memengaruhi proses docking. Dalam kasus seperti itu, bukan hanya struktur yang paling mungkin yang penting, tetapi juga konformasi masuk akal lainnya, karena hal itu dapat memengaruhi hasil dan memberikan pemahaman yang lebih komprehensif tentang interaksi molekuler.
Untuk mengatasi masalah ini dan mengomunikasikan ketidakpastian terkait, bidang UAV telah muncul. Ini berfokus pada mengintegrasikan data dan ketidakpastiannya ke dalam jalur komputasi dan visualisasi terpadu, yang pada akhirnya membantu pengguna membuat keputusan yang lebih tepat. Ini sangat penting karena visualisasi cenderung menunjukkan tingkat akurasi yang jarang ada dalam data [ JS03 ]. Namun, menggabungkan informasi ketidakpastian ke dalam visualisasi menghadirkan tantangannya sendiri. Ini cenderung meningkatkan kompleksitas visual, yang berpotensi menghalangi persepsi data inti [ BHJ*14 ]. Dalam konteks struktur biomolekuler, UV telah menjadi bidang penelitian aktif selama lebih dari 30 tahun, dengan beberapa contoh paling awal ditunjukkan pada Gambar 2 . Meskipun demikian, survei yang ada berfokus pada aspek spesifik visualisasi molekuler atau UV secara umum, tanpa ada survei yang membahas persimpangan unik dari kedua topik ini.

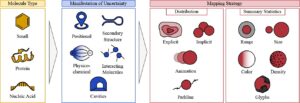
Untuk menyusun dan memberikan gambaran umum tentang karya-karya tentang UAV struktur biomolekuler, kami memperkenalkan skema klasifikasi (Gambar 1 ) yang mengkategorikan pendekatan berdasarkan jenis molekul, manifestasi ketidakpastian, dan pemetaan ketidakpastian. Klasifikasi ini memungkinkan pemahaman yang lebih baik tentang berbagai teknik dan membantu mengidentifikasi tren dan kesenjangan dalam metodologi saat ini. Misalnya, terdapat kekurangan yang signifikan dari teknik UAV untuk data skala besar, meskipun kumpulan data struktural tumbuh pesat. Dengan mengidentifikasi kesenjangan tersebut, kami menyoroti peluang yang menjanjikan untuk memajukan bidang ini.
Kontribusi utama survei kami dapat diringkas sebagai berikut:
- Taksonomi untuk UAV struktur biomolekuler.
- Gambaran menyeluruh penelitian yang ada.
- Identifikasi pola dan kesenjangan dalam metode saat ini.
- Rekomendasi untuk bidang penelitian masa depan yang menjanjikan.
Laporan ini disusun sebagai berikut: Pertama, kami membahas survei terkait (Bagian 2). Selanjutnya, kami memperkenalkan latar belakang yang diperlukan tentang visualisasi struktur molekuler (Bagian 3), dan UAV (Bagian 4). Kemudian, kami membahas cakupan makalah ini secara lebih rinci dan menyajikan taksonomi yang kami usulkan untuk pendekatan visualisasi yang sadar ketidakpastian untuk struktur biomolekuler (Bagian 5). Bagian 6, Bagian 7, dan Bagian 8 kemudian menjelaskan pendekatan untuk molekul kecil, protein, dan asam nukleat. Setelah itu, kami membahas pendekatan yang ada (Bagian 9) dan arah penelitian mendatang (Bagian 10) sebelum kami menyimpulkan laporan ini di Bagian 11.
2. Pekerjaan Terkait
Meskipun survei tentang UV dan visualisasi biomolekuler masing-masing beraneka ragam, survei komprehensif tentang persimpangan kedua area tersebut masih belum ada dalam literatur. Survei tentang visualisasi biologis atau biomolekuler sering kali mengakui adanya ketidakpastian tetapi biasanya membahasnya dalam lingkup yang terbatas, sering kali hanya berfokus pada aspek-aspek individual. Kozlíková dkk. [ KKF*17 ] memberikan gambaran umum yang luas tentang teknik-teknik terkini dalam visualisasi struktur biomolekuler. Laporan mereka mencakup bagian tentang visualisasi dinamika molekuler, yang mencakup metode untuk mengilustrasikan fleksibilitas biomolekuler dan representasi volumetrik.
Krone et al. [ KKL*16 ] mengulas teknik untuk memvisualisasikan rongga biomolekuler, juga membahas beberapa sumber ketidakpastian yang melekat pada bidang ini. Mereka menyoroti tantangan dalam memvisualisasikan rongga dinamis, yang berfluktuasi seiring waktu karena struktur molekuler pada dasarnya dinamis. Selain itu, penulis menekankan perlunya alat visualisasi yang dapat membandingkan dan mengevaluasi berbagai metode deteksi rongga, mencatat perbedaan yang signifikan—hingga 200%—dalam pengukuran volume di seluruh alat. Meskipun ada tantangan ini, hanya beberapa alat yang saat ini dapat menghitung ketidakpastian dalam pengukurannya. Meningkatnya ukuran dan kompleksitas data mendorong perlunya pendekatan visualisasi multiskala. Survei tahun 2019 oleh Miao et al. [ MKK*19 ] mengeksplorasi metode visualisasi molekuler multiskala ini dan menekankan pentingnya visualisasi ikhtisar. Untuk tujuan ini, teknik UV yang sangat abstrak dan/atau agregat ditampilkan dalam tinjauan mereka. Tinjauan terbaru visualisasi biomolekuler oleh Li dan Wei [ LW24 ] mencakup bagian singkat tentang UV posisional, yang menguraikan penyandian umum dan beberapa teknik yang lebih baru.
Karena simulasi dinamika molekul (MD) menghitung sifat dinamis molekul, topik ini terkait erat dengan fleksibilitas molekul dan ketidakpastian posisi. Belghit dkk. [ BSD*24 ] meninjau visualisasi lintasan MD, sementara Corey dkk. [ CBC23 ] secara khusus meninjau visualisasi simulasi MD untuk sistem membran.
Pada tahun 1997, Pang dkk. [ PWL97 ] mengembangkan skema klasifikasi untuk pendekatan UV awal. Skema ini menggabungkan properti data dan visualisasi itu sendiri, seperti dimensionalitasnya dan sifat diskrit atau kontinu. Selain itu, mereka mengkategorikan pendekatan menurut cara mereka menambahkan informasi ketidakpastian ke visualisasi. Mereka mengidentifikasi metode: glif, menambahkan/memodifikasi geometri, memodifikasi atribut, animasi, sonifikasi, dan psikovisual. Brodlie dkk. [ BAOL12 ] membahas alasan mengapa UV menjadi masalah yang sulit. Salah satu alasan utamanya adalah kompleksitas ketidakpastian itu sendiri, bahkan terminologi ketidakpastian sering kali tidak jelas. Untuk UV biomolekuler, misalnya, ketidakpastian sering kali digunakan secara bergantian dengan kesalahan, presisi, atau fleksibilitas. Kesulitan lainnya adalah bahwa ketidakpastian menambahkan dimensi lain ke visualisasi. Untuk data satu atau dua dimensi, hal ini membuat visualisasi lebih kompleks tetapi relatif mudah dipecahkan. Sebagian besar visualisasi struktur biomolekuler pada dasarnya tiga dimensi. Dengan demikian, menambahkan dimensi lain—ketidakpastian—menjadi lebih menantang. Selain itu, mereka secara khusus menekankan perbedaan antara visualisasi ketidakpastian dan ketidakpastian visualisasi : Penelitian biasanya mempertimbangkan visualisasi ketidakpastian dalam kumpulan data. Namun, visualisasi itu sendiri—melalui penyaringan, pemetaan, dan rendering—menambahkan lapisan ketidakpastian lainnya. Bonneau dkk. [ BHJ*14 ] secara formal menggambarkan ketidakpastian dan membahas UV dalam beberapa bidang seperti visualisasi medis atau cuaca dan iklim.
Visualisasi ansambel terkait erat dengan visualisasi ketidakpastian. Faktanya, metafora umum untuk menunjukkan ketidakpastian dalam struktur biomolekuler adalah visualisasi ansambel. Survei visualisasi ansambel oleh Wang et al. [ WHLS19 ] tidak secara eksplisit membahas struktur biomolekuler; namun, beberapa teknik untuk memvisualisasikan ansambel permukaan atau volume saat ini diterapkan pada struktur biomolekuler atau dapat diadaptasi untuk tujuan ini.
Padilla et al. [ PKH21 ] baru-baru ini menguraikan praktik terbaik untuk UV, membahas teori kognitif yang menjelaskan bagaimana metode ini memengaruhi penilaian pemirsa. Mereka terutama memeriksa UV berdimensi lebih rendah (1D, 2D), termasuk bilah kesalahan, larik ikon, dan plot ansambel 2D. Dalam survei mereka tahun 2006 tentang UV, Griethe dan Schumann [ GS06 ] menyoroti kelangkaan studi kegunaan sebagai kesenjangan utama di bidang tersebut. Lebih dari satu dekade kemudian, Hullman et al. [ HQC*19 ] melakukan survei mendalam tentang evaluasi UV, mengidentifikasi beberapa masalah yang belum terselesaikan. Khususnya, mereka mengamati bias penelitian yang lazim terhadap penilaian akurasi kinerja atas kualitas keputusan. Mereka mengusulkan pedoman untuk mengadopsi metode evaluasi yang transparan dan valid.
Weiskopf [ Wei22 ] membahas UV dan mengilustrasikan konsep umum dengan contoh visualisasi data biologis. Akan tetapi, makalahnya bukanlah survei sistematis dan karena berfokus pada data biologis secara umum, hanya beberapa visualisasi struktur molekul yang ditampilkan. Oleh karena itu, tinjauan menyeluruh tentang ketidakpastian dalam visualisasi struktur biomolekuler masih belum ada dalam literatur.
3. Visualisasi Struktur Molekul
Di bagian ini, kami menyediakan tinjauan singkat tentang visualisasi struktur molekuler. Kami mulai dengan ringkasan biomolekul (Bagian 3.1), diikuti dengan uraian berbagai metode untuk memperoleh data struktur biomolekuler (Bagian 3.2). Terakhir, kami membahas model representasi molekuler (Bagian 3.3). Kami hanya menyediakan tinjauan singkat tentang topik-topik ini; untuk informasi lebih mendalam tentang biomolekul dan perolehan data biomolekuler, silakan lihat Nelson dan Cox [ NC21 ], Berg et al. [ BTS10 ], dan Alberts et al. [ AHJ*22 ]. Informasi lebih lanjut tentang model representasi dan visualisasi dapat ditemukan dalam laporan oleh Kozlíková et al. [ KKF*17 ].
3.1. Ringkasan Singkat Biomolekul
Biomolekul sangat penting bagi struktur, fungsi, dan regulasi organisme hidup. Biomolekul merupakan blok pembangun kehidupan dan memainkan peran penting dalam berbagai proses biologis. Bahan utama biomolekul adalah karbon, elemen yang sangat serbaguna yang dapat membentuk berbagai ikatan dengan dirinya sendiri dan elemen lain seperti hidrogen, oksigen, dan nitrogen. Biomolekul dapat diklasifikasikan menjadi dua kelompok utama: molekul kecil dan makromolekul.
Molekul kecil Gambar memiliki berat molekul yang relatif rendah, sangat beragam, dan memiliki banyak fungsi. Misalnya, molekul kecil dapat berfungsi sebagai sumber energi, molekul pemberi sinyal, dan blok penyusun makromolekul yang lebih besar. Meskipun ada banyak jenis molekul kecil, empat kelas utamanya adalah gula, asam lemak, nukleotida, dan asam amino. Molekul-molekul ini membentuk dasar bagi makromolekul yang lebih besar: Karbohidrat yang lebih besar seperti polisakarida dan oligosakarida terbentuk dari molekul gula yang lebih kecil; asam lemak membentuk lemak dan lipid membran; nukleotida merupakan blok penyusun asam nukleat; dan asam amino bergabung membentuk protein.
Karbohidrat berfungsi sebagai sumber energi utama, terutama dalam bentuk gula seperti glukosa. Karbohidrat memberikan dukungan struktural yang penting, terutama pada dinding sel tumbuhan, di mana selulosa memainkan peran penting. Selain itu, karbohidrat berperan dalam pensinyalan seluler, bertindak sebagai molekul pengenalan pada permukaan sel, dan memengaruhi interaksi antara sel dan lingkungannya.
Lipid tidak larut dalam air, penting untuk penyimpanan energi, dan membentuk membran biologis. Lipid membran memiliki kepala hidrofilik dan ekor hidrofobik, yang memungkinkannya membentuk lapisan ganda. Kepala hidrofiliknya menghadap ke luar ke arah air, sedangkan ekor hidrofobiknya menghadap ke dalam, menjauhi air. Struktur lapisan ganda ini membentuk fondasi membran biologis, yang bertindak sebagai penghalang selektif yang memisahkan sel dari lingkungan sekitarnya. Selain peran strukturalnya, beberapa lipid, seperti hormon atau kofaktor enzim, juga berpartisipasi aktif dalam metabolisme.
Asam Nukleat Gambar seperti DNA dan RNA, berperan penting dalam penyimpanan dan penyaluran informasi genetik. DNA mengandung instruksi genetik yang menentukan karakteristik suatu organisme. Di sisi lain, RNA memainkan peran penting dalam menerjemahkan instruksi genetik ini menjadi protein fungsional. Selain itu, beberapa jenis molekul RNA berfungsi dalam berbagai peran regulasi dan katalitik, bertindak sebagai pembawa pesan atau katalis dalam proses biologis. Struktur asam nukleat umumnya dijelaskan dalam empat tingkatan: primer, sekunder, tersier, dan kuartener. Struktur primer asam nukleat mengacu pada urutan linier nukleotida yang dihubungkan oleh ikatan fosfodiester. Setiap nukleotida terdiri dari gula, gugus fosfat, dan basa nitrogen. Struktur sekunder asam nukleat mengacu pada pola pelipatan lokal rantai polinukleotida. Dalam DNA, hal ini terlihat jelas dalam heliks ganda, struktur spiral tempat dua untai nukleotida yang saling melengkapi membentuk ikatan hidrogen antara pasangan basa yang saling melengkapi. Dalam RNA, struktur sekunder mencakup daerah untai tunggal dan ganda. Pada daerah untai ganda, pasangan basa komplementer membentuk ikatan hidrogen. Ciri khas struktur sekunder RNA adalah simpul jepit rambut, tempat segmen RNA untai tunggal terlipat kembali pada dirinya sendiri untuk membentuk batang untai ganda. Batang ini disatukan oleh pasangan basa komplementer, sementara lipatan menciptakan simpul untai tunggal di ujung batang lainnya. Gambar 3a menunjukkan contoh simpul jepit rambut. Struktur tersier asam nukleat didefinisikan oleh koordinat atom molekul dan mengacu pada bentuk tiga dimensi keseluruhan yang dihasilkan dari pelipatan dan pelintiran lebih lanjut dari struktur sekunder. Struktur kuartener mengacu pada organisasi tingkat tinggi molekul asam nukleat, seperti susunan beberapa subunit RNA menjadi kompleks fungsional.

Protein Gambar menjalankan berbagai fungsi biologis penting. Enzim mengkatalisis reaksi kimia seperti metabolisme dan replikasi DNA. Protein lain menyediakan dukungan struktural dalam sel dan jaringan. Dalam transportasi molekuler, mereka memastikan pergerakan molekul melintasi membran. Selain itu, protein memainkan peran kunci dalam respons imun dengan mengenali dan menetralkan patogen. Keragaman mereka memungkinkan mereka untuk melakukan berbagai peran ini. Protein tersusun dari rantai panjang asam amino, dan urutan spesifik asam amino ini menentukan bagaimana protein terlipat dan berfungsi. Seperti asam nukleat, protein dijelaskan oleh struktur primer, sekunder, tersier, dan kuartenernya. Struktur primer mengacu pada urutan linier asam amino dalam rantai polipeptida. Struktur sekunder melibatkan pelipatan rantai polipeptida terlokalisasi menjadi pola teratur, yang distabilkan oleh ikatan hidrogen. Heliks alfa dan lembaran beta adalah pola yang paling umum. Struktur tersier menjelaskan bagaimana seluruh rantai polipeptida terlipat menjadi bentuk tiga dimensi. Ketika beberapa polipeptida bergabung untuk membentuk kompleks protein, susunannya dijelaskan oleh struktur kuartener. Penataan spasial protein sangat penting bagi fungsinya. Bahkan perubahan kecil dalam strukturnya dapat menyebabkan disfungsi dan penyakit.
3.2. Akuisisi Data
Data molekuler struktural paling sering diperoleh secara in vitro menggunakan salah satu dari tiga teknik: kristalografi sinar-X, NMR, atau mikroskopi elektron kriogenik (cryo-EM).
Sebagian besar data struktural yang tersedia saat ini diperoleh melalui kristalografi sinar-X. Teknik ini memberikan detail resolusi tinggi dan efektif untuk molekul kecil dan makromolekul. Dalam proses ini, berkas sinar-X melewati kristal molekuler, dan pola difraksi yang dihasilkan digunakan untuk menghasilkan peta kerapatan elektron. Peta kerapatan elektron ini menggambarkan distribusi probabilitas posisi elektron. Untuk menghasilkan data koordinat atom, peta dianalisis, dan atom diposisikan di dalamnya. Persyaratan utama untuk metode ini adalah membuat kristal molekuler. Kondisi kristalisasi perlu disesuaikan dengan hati-hati untuk setiap molekul, dan diperlukan sejumlah besar sampel yang dimurnikan.
NMR digunakan untuk mempelajari molekul dalam larutan. Sampel ditempatkan dalam medan magnet yang kuat, di mana pulsa radiasi elektromagnetik mengubah putaran inti atom. Sinyal yang dihasilkan, yang dikenal sebagai pergeseran kimia, dianalisis untuk menetapkan kemungkinan struktur 3D. NMR terutama digunakan untuk menguraikan struktur molekul yang lebih kecil.
Cryo-EM digunakan untuk menganalisis struktur yang sangat besar, seperti kompleks protein besar, yang seringkali sulit dikristalkan dan terlalu besar untuk NMR. Lapisan tipis sampel dibekukan dengan cepat untuk mempertahankan bentuk molekul. Berkas elektron diarahkan melalui lapisan tersebut, dan detektor menangkap elektron yang ditransmisikan untuk menghasilkan gambar 2D. Kumpulan molekul yang berorientasi serupa ditumpangkan untuk meningkatkan rasio sinyal terhadap derau, dan tampilan ini digabungkan menjadi peta kerapatan elektron 3D. Untuk molekul yang relatif stabil, resolusi cryo-EM dapat mendekati resolusi kristalografi sinar-X.
Teknik in vitro ini dilengkapi dengan teknik in silico seperti pemodelan dan simulasi molekuler. Sementara teknik in vitro sangat diperlukan untuk memvalidasi temuan dan memberikan bukti eksperimental langsung, teknik in silico mengisi celah dengan menawarkan wawasan tingkat atom, berkelanjutan, dan terkendali lingkungan yang sulit atau tidak mungkin dicapai hanya secara in vitro. Simulasi MD umumnya digunakan untuk memodelkan gerakan molekuler, mensimulasikan pergerakan atom dari waktu ke waktu. Medan gaya menggambarkan interaksi antara atom-atom individual, memandu bagaimana mereka bergerak. Posisi atom diperbarui pada setiap langkah waktu, menghasilkan data lintasan yang melacak evolusi sistem. Ini memungkinkan penyempurnaan data struktural yang dihasilkan secara eksperimental, memodelkan stabilitas molekuler, dan memungkinkan simulasi interaksi antara beberapa molekul.
Simulasi MD dapat diperluas dengan beberapa cara untuk meningkatkan kegunaannya dalam mempelajari proses biomolekuler. Misalnya, gaya simulasi tertentu, seperti yang digunakan dalam pengambilan sampel payung atau metadinamika, dapat diterapkan untuk mempelajari proses kompleks seperti pelipatan dan penguraian protein dengan mendorong eksplorasi konformasi langka dan keadaan transisi. Selain itu, teknik berbutir kasar mengurangi kompleksitas sistem besar dengan menyederhanakan detail atom, memungkinkan simulasi sistem biomolekuler yang lebih besar dalam skala waktu yang lebih lama. Perluasan hebat lainnya adalah integrasi dengan mekanika kuantum (QM), di mana pendekatan mekanika kuantum/mekanika molekuler (QM/MM) memungkinkan pemodelan akurat situs reaktif atau reaksi kimia dalam lingkungan molekuler yang lebih besar, memberikan wawasan yang lebih tepat tentang mekanisme enzimatik atau interaksi obat.
Pendekatan alternatif adalah metode Monte Carlo (MC). Metode MC adalah teknik komputasi yang mengeksplorasi konfigurasi molekuler melalui pengambilan sampel acak daripada evolusi waktu berkelanjutan. Alih-alih menghitung gaya dan mengintegrasikan persamaan gerak seperti dalam MD, MC secara acak memilih konfigurasi baru, yang diterima atau ditolak berdasarkan kriteria seperti algoritma Metropolis, yang memastikan bahwa keadaan energi rendah lebih mungkin untuk diambil sampelnya. MC sangat berguna untuk mempelajari sifat kesetimbangan, pengambilan sampel konformasi, dan sifat termodinamika biomolekul. Metode ini dapat diterapkan pada struktur yang direpresentasikan pada detail atom atau dengan teknik berbutir kasar, tergantung pada tingkat detail yang dibutuhkan. Selain itu, MC sering dikombinasikan dengan teknik pengambilan sampel yang ditingkatkan, seperti pengambilan sampel payung, untuk mengatasi hambatan energi dan mengeksplorasi konfigurasi langka yang mungkin tidak dapat diakses.
Teknik lain yang kurang umum adalah analisis mode normal (NMA). NMA menyediakan metode untuk memeriksa gerakan kolektif yang besar dalam biomolekul. Dengan memodelkan molekul sebagai sekumpulan massa titik yang dihubungkan oleh pegas harmonik, NMA mengidentifikasi gerakan frekuensi rendah, yang biasanya berhubungan dengan perubahan fungsional dalam struktur protein. Pendekatan ini efisien secara komputasi, menjadikannya alat yang berguna untuk mempelajari dinamika konformasi dalam skala luas, sering kali dikombinasikan dengan metode yang lebih rinci seperti simulasi MD dan MC.
3.3. Model Representasi Molekuler
Ada beberapa opsi untuk membuat representasi molekuler dari data yang diperoleh. Di bagian ini, kami mengikuti klasifikasi Kozlíková et al. [ KKF*17 ] yang mengidentifikasi tiga kategori utama: Model atomistik, model abstrak atau ilustratif, dan model level of detail (LOD). Model atomistik memberikan tampilan model yang sangat rinci dengan menampilkan atom atau ikatan individual. Model tersebut selanjutnya dapat dipisahkan menjadi model yang berpusat pada ikatan dan model permukaan. Model yang berpusat pada ikatan umum dalam kimia dan untuk molekul yang lebih kecil. Model tersebut menekankan hubungan antara atom-atom individual. Representasi paling dasar menggunakan garis untuk menggambarkan ikatan, sementara varian yang lebih canggih, model licorice atau stick, menggunakan silinder sebagai pengganti garis. Ketika bola juga digunakan untuk merepresentasikan atom, penggambaran yang dihasilkan disebut model bola dan tongkat. Model yang berpusat pada ikatan dapat dihitung secara efisien pada GPU menggunakan teknik raycasting dan imposter-based. Gambar 2a dan Gambar 2b adalah contoh untuk representasi yang berpusat pada ikatan.
Model permukaan menggambarkan susunan spasial atom sebagai permukaan kontinu. Representasi molekuler paling sederhana adalah model pengisian-ruang, atau model calotte, di mana atom digambarkan sebagai bola dengan ukuran yang proporsional dengan jari-jari atomnya (lihat Gambar 4a ). Meskipun representasi ini sendiri bukanlah permukaan kontinu, permukaan dapat dihasilkan dengan menggunakan permukaan luar gabungan semua bola. Ini dilakukan, misalnya, untuk permukaan van der Waals (vdW) [ Ric77 ], di mana ukuran bola sesuai dengan jari-jari vdW atom (lihat Gambar 4b ). Permukaan ini adalah dasar untuk beberapa representasi lainnya. Permukaan yang dapat diakses pelarut (SAS) [ LR71 ] dihitung dengan menggulirkan bola probe (biasanya seukuran molekul air) di atas permukaan vdW. Bagian tengah bola probe dilacak dan menandai SAS, menghasilkan permukaan yang lebih halus dengan lebih sedikit diskontinuitas daripada permukaan vdW. Ini menyoroti daerah yang dapat diakses oleh molekul yang berinteraksi. Namun, pendekatan ini mengembang permukaan molekuler, karena SAS menyertakan radius bola probe. Permukaan yang dikecualikan pelarut (SES) [ Ric77 ] mengatasi masalah ini dengan melacak titik bola probe yang paling dekat dengan atom (lihat Gambar 4c ). Metode ini merepresentasikan volume molekuler dengan lebih tepat. Representasi permukaan yang kurang umum mencakup permukaan kulit molekuler (MSS) [ Ede99 ], yang secara terus-menerus dapat dibedakan tetapi tidak memiliki dasar biofisik dan secara komputasi rumit. Contoh lain adalah permukaan yang dikecualikan ligan (LES) [ LBH14 ], yang memperluas konsep SES dengan menggabungkan geometri permukaan vdW ligan. Model permukaan konvolusi [ Bli82 ] menawarkan pendekatan visualisasi alternatif dengan memadukan potensi atom menggunakan kernel konvolusi. Mereka sering kali efisien secara komputasi, dengan beberapa menyediakan salah satu algoritma ekstraksi permukaan tercepat yang tersedia.
Model abstrak dan ilustratif tidak menggambarkan atom-atom secara langsung. Model-model ini khususnya berguna untuk merepresentasikan makromolekul besar. Model-model ini sering kali diturunkan dari model permukaan yang dibahas sebelumnya. Alih-alih berfokus pada atom-atom secara individual, model-model ini memperlakukan kelompok atom—seperti blok-blok penyusun molekul seperti asam amino—sebagai unit terkecil untuk pembuatan permukaan. Permukaan konvolusi bahkan dapat langsung diadaptasi dengan menyesuaikan parameter-parameternya untuk menghasilkan visualisasi yang lebih kasar dan lebih abstrak. Metode-metode abstraksi semacam itu meningkatkan efisiensi rendering dan mengurangi kekacauan visual.
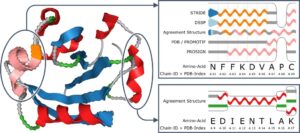
Model ilustratif menyorot fitur-fitur utama dalam data yang sulit dibedakan dengan tampilan atomistik. Model ini sering digunakan untuk menggambarkan struktur sekunder protein. Misalnya, representasi kartun [ Ric81 ] menunjukkan heliks alfa sebagai pita, lembaran beta sebagai anak panah, dan lingkaran sebagai garis. Ada beberapa varian representasi kartun, seperti menggunakan silinder untuk merepresentasikan lembaran beta. Visualisasi struktur pada Gambar 5 menggambarkan varian representasi kartun. Metode ilustratif lainnya merepresentasikan tulang punggung sebagai tabung kontinu. Representasi heliks ganda DNA yang terkenal adalah contoh lain dari visualisasi ilustratif.
4. Visualisasi yang Menyadari Ketidakpastian
Visualisasi ketidakpastian (UV) berfokus pada representasi eksplisit kesalahan atau ketidakpastian dalam data. Visualisasi yang menyadari ketidakpastian (UAV) membangun hal ini dengan mengintegrasikan visualisasi ketidakpastian secara langsung dengan data itu sendiri, memastikan keduanya terwakili secara efektif [ GSWS21 ]. Di sini, kami fokus pada pendekatan terakhir, di mana data dan ketidakpastiannya divisualisasikan bersama. Namun, visualisasi tersebut sering kali masih disebut sebagai UV. Di bagian ini, kami menguraikan sumber ketidakpastian potensial (Bagian 4.1), membahas bagaimana ini memetakan ke data biomolekuler (Bagian 4.2), memperkenalkan strategi pemetaan umum untuk memvisualisasikan ketidakpastian (Bagian 4.3), dan membahas secara spesifik visualisasi ketidakpastian untuk data biomolekuler dan kesamaan dengan domain lain (Bagian 4.4).
4.1. Sumber Ketidakpastian
Dalam konteks visualisasi, sumber ketidakpastian biasanya dibahas dalam kaitannya dengan masing-masing tahap alur visualisasi [ PWL97 , BAOL12 , BHJ*14 ]. Untuk tahap akuisisi data, sumber utama ketidakpastian adalah variasi statistik. Pengukuran eksperimental sering menunjukkan fluktuasi; pengukuran berulang dengan kuantitas yang sama dalam kondisi yang sama menghasilkan hasil yang sedikit berbeda [ GMR*23 ]. Ada juga ketidakpastian pengambilan sampel: dalam survei statistik, bahkan ketika sampel representatif, sampel yang berbeda dapat menghasilkan hasil yang sedikit berbeda karena variabilitas alami dalam populasi [ KF10 ]. Di luar variasi statistik, ketidakpastian juga berasal dari bias sistematis, misalnya, pengambilan sampel yang tidak representatif dapat menghasilkan kumpulan data yang bias atau tidak lengkap ketika teknik pengambilan sampel yang tidak tepat digunakan [ GMR*23 ]. Sumber ketidakpastian lainnya adalah ketidaklengkapan data [ GMR*23 ] (yaitu, titik data yang hilang atau tidak tersedia). Selain itu, resolusi instrumen yang terbatas menimbulkan ketidakpastian karena keterbatasan inheren sensor yang menangkap fenomena fisik [ GMR*23 ]. Setiap sensor memiliki resolusi dan jangkauan tertentu, yang membatasi ketepatan pengukuran. Dalam data yang diperoleh dari simulasi atau pemodelan, ketidakpastian muncul karena keterbatasan daya komputasi dan pengetahuan. Ketidaklengkapan model merupakan masalah umum, karena tidak ada model yang dapat sepenuhnya mewakili fenomena dunia nyata, dan penyederhanaan atau asumsi sering kali diperkenalkan selama konstruksi model [ WSC21 ]. Ketidakpastian parameter juga memengaruhi model komputasi, karena mengidentifikasi parameter optimal merupakan tantangan dan sering kali memerlukan penentuan eksperimental [ GMR*23 ].
Ketidakpastian yang muncul selama akuisisi data biasanya merupakan ketidakpastian utama yang divisualisasikan. Namun, ketidakpastian tambahan dapat muncul selama tahap pemrosesan, pemodelan, dan rendering. Kami hanya membahasnya secara singkat di sini dan merujuk ke Brodlie et al. [ BAOL12 ] untuk pembahasan yang lebih terperinci. Selama pemrosesan data, misalnya, data sering dihaluskan atau diinterpolasi. Dengan menginterpolasi titik data (seperti menggambar kurva melalui titik data diskrit), nilai pasti pada titik tertentu hanya dapat diperkirakan. Dalam tahap pemodelan, representasi data yang dipilih tunduk pada ketidakpastian. Ketidakpastian mungkin muncul karena perkiraan atau penyederhanaan, seperti merepresentasikan fenomena kontinu dengan model diskrit (misalnya, menggunakan jaring untuk merepresentasikan permukaan halus). Selama tahap rendering, ketidakpastian lebih lanjut muncul. Rasterisasi, misalnya, secara inheren diskrit dan memiliki resolusi terbatas; akibatnya, detail kecil mungkin hilang. Atau, teknik ray tracing yang berbeda dapat menghasilkan hasil yang berbeda tergantung pada algoritma atau pengaturan, yang menambah lebih banyak ketidakpastian pada visualisasi akhir. Terakhir, persepsi pemirsa menimbulkan sumber ketidakpastian lain [ GMR*23 ]. Faktor-faktor seperti pengalaman sebelumnya, kekurangan penglihatan, dan literasi visualisasi dapat memengaruhi cara individu menginterpretasikan visualisasi. Keterbatasan visual, bias kognitif, dan faktor kontekstual semuanya dapat berkontribusi pada interpretasi yang berbeda-beda, sehingga sulit untuk memastikan komunikasi ketidakpastian yang akurat.
4.2 Ketidakpastian dalam Biomolekul
Dalam data struktur biomolekuler, seperti di banyak bidang UV, fokus utamanya adalah pada ketidakpastian yang muncul selama tahap akuisisi data. Ini tidak berarti bahwa ketidakpastian tidak ada atau tidak relevan dalam tahap lain dari alur visualisasi. Namun, sejauh pengetahuan kami, kuantifikasi ketidakpastian untuk tahap-tahap selanjutnya ini belum dieksplorasi. Ketidakpastian yang muncul selama tahap-tahap selanjutnya kemungkinan serupa dengan ketidakpastian yang ditemukan dalam UV umum.
Dalam tahap akuisisi data, salah satu ketidakpastian yang paling kritis adalah ketidakpastian posisi. Posisi atom atau bentuk molekul memainkan peran penting dalam banyak analisis, seperti simulasi docking atau penyelarasan struktural. Bahkan ketika kuantitas yang diinginkan (QOI) tidak secara eksplisit bersifat posisi, ketidakpastian dalam posisi atom masih menyebar ke dan memengaruhi QOI [ RCBB19 ]. Sumber pasti ketidakpastian posisi bervariasi dengan metode akuisisi data. Di bawah ini, kami menguraikan beberapa sumber penting ketidakpastian posisi untuk struktur biomolekuler.
Untuk data in vitro, ketidakpastian seperti fluktuasi statistik, variabilitas lingkungan, dan keterbatasan dalam resolusi dan jangkauan instrumental memengaruhi semua teknik akuisisi struktur. Selain itu, setiap metode akuisisi memperkenalkan sumber ketidakpastian tertentu: Dalam kristalografi sinar-X, kristal molekuler harus diproduksi untuk proses pencitraan; dengan demikian, konformasi struktur dalam keadaan yang lebih alami—seperti dalam larutan—tetap tidak pasti. Ketidakpastian pemodelan muncul selama pembuatan peta kerapatan elektron dari pola difraksi. Setelah itu, model struktural dipasang ke dalam peta kerapatan elektron. Proses pemasangan ini biasanya memilih struktur berdasarkan kecocokan kerapatan terbaik dan kendala tambahan, seperti interaksi biofisik, urutan asam amino, dan struktur sekunder atau tersier [ RCBB19 ]. Untuk menilai kualitas kecocokan struktural, beberapa metrik tersedia. Metrik umum adalah faktor-b, yang mengukur perpindahan atau getaran atom individu di sekitar posisi rata-ratanya [ MDC24 ]. Ini berasal dari redaman hamburan sinar-X. Dalam spektroskopi NMR, urutan asam amino harus diketahui untuk memperoleh struktur dari spektrum NMR. Ketidakpastian dalam data masukan ini menyebar ke seluruh proses. Lebih jauh, beberapa struktur sering kali memenuhi batasan dengan baik, menghasilkan himpunan struktur yang secara implisit mengodekan distribusi kemungkinan konformasi. Untuk cryo-EM, pemodelan struktural melibatkan jalur yang panjang. Ribuan gambar molekuler harus diperoleh, difilter menurut orientasinya, dan ditumpangkan untuk meningkatkan rasio sinyal terhadap derau. Gambar yang diproses ini kemudian digabungkan menjadi peta kerapatan 3D. Pada setiap tahap jalur ini, ketidakpastian menyebar dari data awal, dan ketidakpastian tambahan diperkenalkan. Akhirnya, seperti halnya kristalografi sinar-X, struktur harus disesuaikan dengan peta kerapatan yang dihasilkan. Berbagai sumber ketidakpastian ini sering kali tersedia dengan mudah. Basis data struktural seperti RCSB Protein Data Bank ( rcsb.org ) (PDB) [ BWF*00 ] menyediakan faktor-b, konformasi alternatif, dan peta kerapatan elektron, yang berfungsi sebagai indikator langsung ketidakpastian pengukuran.
Untuk akuisisi data in silico, struktur yang diselesaikan secara eksperimental biasanya digunakan sebagai data input, yang berarti ketidakpastian dari akuisisi struktur menyebar melalui jalur pipa. Ketidakpastian tambahan muncul selama proses simulasi. Misalnya, simulasi MD sangat sensitif terhadap kondisi awal. Mereka menunjukkan perilaku kacau, menghasilkan penyimpangan yang tampaknya acak yang membuat pengamatan tunggal sebagian besar tidak dapat direproduksi. Menghasilkan ansambel alih-alih mengandalkan simulasi tunggal dapat meningkatkan keandalan dan memungkinkan estimasi ketidakpastian yang dapat diukur [ WSC21 ]. Pilihan parameter sangat penting tidak hanya dalam simulasi tetapi juga dalam tugas komputasi lainnya. Misalnya, dalam penyelarasan sekuens ganda, prediksi dapat sangat bervariasi tergantung pada parameter seperti biaya gap [ Ham14 ].
Ukuran umum untuk menganalisis jarak antara konformasi dalam suatu ensemble adalah deviasi kuadrat rata-rata (RMSD) [ LGV*20 ]. Ini menghitung perbedaan antara posisi atom dalam struktur individual dan struktur referensi (seringkali struktur rata-rata ensemble). Jika RMSD dihitung selama deret waktu yang berpusat di sekitar struktur rata-rata yang relatif stabil, itu disebut sebagai RMSF. Data simulasi juga dipengaruhi oleh kesalahan sistematis, seperti ketidaksempurnaan dalam desain, eksekusi, atau analisis. Contoh spesifik dari desain yang tidak sempurna dalam MD adalah penggunaan medan gaya protein tertentu yang cenderung mendukung jenis struktur sekunder tertentu. Mengenali bias ini telah menyebabkan upaya untuk mengembangkan medan gaya yang lebih baik yang bertujuan untuk mengurangi atau menghilangkan kesalahan sistematis ini [ WSC21 ]. Kesalahan lain termasuk formulasi model yang disederhanakan, perkiraan algoritmik seperti representasi diskrit dari model kontinu, integrator numerik, dan akumulasi kesalahan pembulatan [ RCBB19 , WSC21 ].
Seperti yang dibahas sebelumnya, menghasilkan data biomolekuler untuk visualisasi struktur adalah proses yang panjang, yang secara inheren membuatnya rentan terhadap akumulasi ketidakpastian pada setiap langkah kemajuan. Ketidakpastian yang diperkenalkan dalam satu langkah menyebar ke seluruh kemajuan, dengan ketidakpastian tambahan yang berpotensi diperkenalkan pada setiap langkah berikutnya. Masalah ini bahkan lebih parah jika QOI tambahan dihitung berdasarkan struktur, yang memperluas jalur pipa dan memperkuat ketidakpastian [ RCBB19 ]. Contoh utama dari hal ini adalah ketidakpastian fisikokimia. Ketidakpastian fisikokimia adalah ketidakpastian dalam sifat fisik dan kimia molekul, seperti distribusi muatan, hidrofobisitas, dan gaya vdW. Sifat-sifat ini sangat penting untuk memahami bagaimana molekul berinteraksi satu sama lain dan lingkungannya. Rasheed et al. [ RCBB19 ] menemukan bahwa ketidakpastian posisi—lebih khusus lagi faktor-b—tampaknya menyebar hanya ke ketidakpastian rendah untuk kuantitas sederhana (misalnya, luas permukaan yang terekspos) tetapi ketidakpastian tinggi untuk kuantitas kompleks seperti energi total.
Tantangan lain dalam pemrosesan lebih lanjut adalah estimasi dalam ruang diskrit berdimensi tinggi, seperti penugasan struktur sekunder atau penyelarasan sekuens. Dalam masalah ini jumlah solusi sangat banyak—berskala secara eksponensial dengan panjang sekuens—menghasilkan probabilitas yang sangat kecil untuk setiap solusi tunggal [ Ham14 ]. Cara yang mungkin untuk membuat perhitungan lebih andal adalah metode ensemble atau probabilitas marginal. Probabilitas marginal adalah probabilitas tanpa syarat yang diperoleh dengan mengintegrasikan probabilitas gabungan atas semua nilai yang mungkin dari variabel lain dalam sistem. Akibatnya, probabilitas marginal ini jauh lebih besar daripada probabilitas solusi individu mana pun [ Ham14 ]. Probabilitas pasangan basa adalah contoh probabilitas marginal dan berfungsi sebagai ukuran ketidakpastian umum untuk struktur sekunder dalam RNA. Ini mengacu pada kemungkinan bahwa dua nukleotida akan membentuk pasangan. Ini menjumlahkan probabilitas semua konformasi di mana kedua basa membentuk pasangan. Konsep serupa dalam penyelarasan sekuens disebut probabilitas pasangan yang selaras.
Sementara ketidakpastian posisi biasanya mudah diakses untuk visualisasi, manifestasi ketidakpastian lainnya sering kali memerlukan inferensi tidak langsung, seperti model konsensus atau skor keyakinan. Mengatasi tantangan ini tetap menjadi bidang penelitian yang sedang berlangsung dalam kuantifikasi dan visualisasi ketidakpastian.
4.3. Strategi Pemetaan
Memvisualisasikan ketidakpastian dalam data sangat penting untuk secara akurat mewakili variabilitas dan keterbatasan informasi yang melekat. Berbagai strategi dapat digunakan untuk memetakan ketidakpastian ini ke visualisasi, masing-masing dengan kekuatannya sendiri tergantung pada jenis data dan audiens serta tugas yang dituju. Dalam bab ini, kami membahas kemungkinan strategi pemetaan untuk ketidakpastian dalam data biomolekuler yang ditemukan dalam literatur. Seperti disebutkan di atas, tantangan utama dalam UV biomolekuler adalah bahwa ketidakpastian biasanya perlu divisualisasikan bersama dengan atribut biokimia lainnya untuk memungkinkan analisis yang komprehensif. Kami mengikuti klasifikasi oleh Weiskopf et al. [ Wei22 ] dan terutama membedakan antara menampilkan distribusi ketidakpastian secara langsung dan memvisualisasikan statistik ringkasan.
4.3.1. Visualisasi distribusi
Visualisasi langsung dari distribusi ketidakpastian Gambardapat memberikan gambaran menyeluruh dari seluruh ruang ketidakpastian. Visualisasi ini khususnya berguna dalam skenario yang mengharuskan pemahaman berbagai kemungkinan. Sementara visualisasi ini menawarkan wawasan terperinci tentang sifat ketidakpastian, visualisasi ini juga meningkatkan kompleksitas visualisasi. Visualisasi distribusi eksplisit merepresentasikan distribusi secara langsung, dengan contoh umum termasuk plot kepadatan, plot titik, histogram, dan plot biola. Metode ini khususnya cocok untuk kumpulan data satu dimensi, karena informasi ketidakpastian sering dipetakan ke sumbu tambahan. Untuk kumpulan data tiga dimensi, seperti struktur molekuler, visualisasi eksplisit biasanya melibatkan rendering volume. Karena sumbu tambahan tidak layak untuk kumpulan data ini, distribusi dirender dengan memodifikasi properti visual, seperti menyesuaikan opasitas berdasarkan kepadatan distribusi.
Pendekatan lain adalah memvisualisasikan distribusi secara implisit Gambardengan mengambil sampel dan menampilkan titik data individual, sering kali melalui superposisi atau penjajaran. Contohnya termasuk diagram sebar, diagram spaghetti, dan kelipatan kecil. Untuk struktur biomolekuler, ini sering dilakukan melalui visualisasi ansambel yang melapisi beberapa konformasi menggunakan bentuk abstrak seperti representasi kartun.
Animasi sangat erat kaitannya dengan pemetaan implisit Gambar. Animasi untuk UV memanfaatkan perubahan temporal untuk menyampaikan dinamika ketidakpastian. Misalnya, titik data dapat berkedip atau berosilasi sedikit dalam wilayah terbatas, yang merepresentasikan ketidakpastian posisi atau nilai. Demikian pula, permukaan dapat mengembang atau menyusut seiring waktu. Plot Hasil Hipotetis [ HRA15 ], yang menganimasikan sampel yang diambil dari distribusi ketidakpastian, adalah contoh UV yang relatif baru. Animasi bergantung pada dinamika visual yang familier tanpa memperkenalkan penyandian baru. Namun, animasi memerlukan waktu sebagai dimensi tambahan, yang dapat mempersulit perolehan gambaran umum data dan dapat membatasi representasi akurat dari distribusi kompleks. Garis lintasan Gambardapat digunakan untuk membuat representasi statis dari animasi. Meskipun garis lintasan secara tradisional tidak dianggap sebagai pemetaan untuk ketidakpastian, garis lintasan menyoroti perilaku dinamis. Dengan melapisi beberapa garis lintasan alternatif untuk fitur yang sama, garis lintasan merupakan bentuk alternatif visualisasi ansambel. Contoh teknik ini dapat dilihat dalam visualisasi ansambel prakiraan untuk jalur siklon tropis [ LPCH19 ].
4.3.2. Memvisualisasikan statistik ringkasan
Bahasa Indonesia: Alternatif untuk menampilkan distribusi ketidakpastian secara langsung adalah visualisasi statistik ringkasan. Visualisasi ini lebih ringkas dan berfokus pada properti spesifik dari distribusi ketidakpastian. Sementara pendekatan ini mengorbankan beberapa detail tentang ketidakpastian, sering kali lebih mudah untuk diintegrasikan ke dalam visualisasi dan berguna jika hanya aspek tertentu dari ketidakpastian yang relevan dengan tugas tersebut. Metode yang tersebar luas untuk merepresentasikan ketidakpastian dalam statistik ringkasan adalah melalui rentang Gambar. Dalam data satu dimensi, ini biasanya dilakukan dengan menggunakan batang kesalahan atau interval kepercayaan. Dalam diagram garis, pita kepercayaan—wilayah di sekitar garis tengah—sering kali digunakan untuk menggambarkan interval ketidakpastian. Untuk data tiga dimensi, rentang ketidakpastian sering kali direpresentasikan sebagai lambung ketidakpastian.
Alternatifnya, variabel visual seperti ukuran Gambar, warna Gambar, dan kepadatan Gambardapat dimodifikasi untuk merepresentasikan ketidakpastian. Misalnya, dalam diagram sebar, titik data dapat dibuat lebih besar ketika ketidakpastian tinggi dan lebih kecil ketika ketidakpastian rendah. Dalam visualisasi struktur biomolekuler, warna sering kali merepresentasikan ukuran ketidakpastian, seperti faktor-b, pada representasi permukaan molekuler. Variasi kepadatan juga dapat menggambarkan ketidakpastian, misalnya, dengan menyesuaikan kepadatan primitif dalam teknik tekstur seperti stippling atau arsir berdasarkan tingkat ketidakpastian. Dalam diagram garis, frekuensi garis putus-putus dalam garis putus-putus dapat ditingkatkan atau dikurangi untuk mencerminkan ketidakpastian. MacEachren dkk. [ MRO*12 ] membahas dan mengevaluasi beberapa variabel visual dan utilitasnya untuk merepresentasikan ketidakpastian. Pendekatan lain melibatkan penggunaan atau modifikasi glif Gambaruntuk menunjukkan ketidakpastian. Misalnya, glif telah digunakan untuk memvisualisasikan medan vektor, dengan atributnya diubah untuk mencerminkan ketidakpastian [ WPL96 ] atau untuk merepresentasikan tensor tak pasti dengan menambahkan hull transparan ke glif [ GRT19 ].
Terakhir, visualisasi terpisah dapat digunakan untuk merepresentasikan ketidakpastian di samping visualisasi data. Pendekatan ini umum dalam sistem UV. Namun, laporan ini berfokus pada UAV dan metode yang mengintegrasikan informasi ketidakpastian secara langsung ke dalam representasi visual data.
4.4. Kekhususan dan kesamaan biomolekuler
Visualisasi ketidakpastian dalam data struktur biomolekuler menantang karena sifatnya yang tiga dimensi dan beragam kemungkinan representasi. Struktur molekul dapat digambarkan menggunakan berbagai representasi, termasuk kartun dan rendering permukaan. Selain itu, data diselidiki pada berbagai tingkat granularitas, mulai dari model kuantum-fisik hingga mesoskala. Selain itu, ensembel, yang menangkap berbagai status konformasi, sangat penting untuk visualisasi struktur biomolekuler. Namun, ada juga kebutuhan untuk menyajikan nilai ketidakpastian skalar. Lebih jauh, visualisasi sering kali mengintegrasikan data ketidakpastian dengan sifat fisikokimia lainnya. Sementara kombinasi tantangan ini khusus untuk struktur biomolekuler, ada juga kesamaan dengan domain lain. Di sini, kami menyoroti dua domain terpilih untuk menunjukkan kesamaan dengan UV biomolekuler.
Contoh domain di mana visualisasi ketidakpastian sangat penting adalah ilmu bumi. Dalam ilmu iklim, UV digunakan secara luas untuk merepresentasikan ketidakpastian dan variabilitas dalam proyeksi model iklim. Data ensemble sangat relevan bagi ilmu bumi dan data biomolekuler. Teknik seperti visualisasi data ensemble dapat mengungkap penyebaran nilai variabel dalam urutan model yang dijalankan, sehingga menghasilkan pemahaman yang lebih baik tentang ketidakpastian model [ PWB*09 ]. Lebih jauh lagi, visualisasi ketidakpastian dapat memengaruhi pengambilan keputusan dalam prakiraan cuaca. Studi menunjukkan bahwa beberapa teknik visualisasi meningkatkan interpretasi dan pemanfaatan ketidakpastian prakiraan [ NJT08 ]. Dalam simulasi laut, Raith et al. [ RSG21 ] mengidentifikasi dan mengukur berbagai ketidakpastian dan menyajikan pendekatan visualisasi yang mengintegrasikan ketidakpastian ini untuk menggambarkan pusaran laut. Pendekatan ini meningkatkan persepsi dinamika laut dengan memfasilitasi penggambaran umum ketidakpastian dalam mendeteksi pusaran, seperti yang ditunjukkan dalam studi kasus Laut Merah. Berbeda dengan data biomolekuler, visualisasi ketidakpastian untuk ilmu bumi sering menampilkan ketidakpastian dalam visualisasi 2D seperti peta.
Dalam visualisasi medis, bagaimanapun, representasi 3D adalah umum: Dalam visualisasi data medis multimodal, beberapa teknik digunakan untuk mengodekan berbagai jenis informasi ke permukaan [ LSBP18 ]. Bentuk permukaan organik di sini memiliki sifat geometris yang mirip dengan beberapa representasi molekuler seperti SES. Lebih jauh lagi, visualisasi medis memadukan data heterogen untuk menjaga kepercayaan pengguna. Selain pengodean berbasis warna, visualisasi data multimodal bergantung pada teknik ilustrasi [ LBSP14 , LP16 , LVPI18 ]. Tantangan dalam menampilkan serangkaian data yang beragam mirip dengan visualisasi struktur biomolekuler, di mana beberapa fitur fisikokimia harus direpresentasikan secara bersamaan. Contoh spesifik visualisasi ilustrasi untuk visualisasi medis multimodal adalah PelVis [ SLK*17 ], di mana visualisasi ilustrasi diterapkan untuk operasi panggul onkologis untuk meminimalkan risiko kerusakan saraf otonom dan komplikasi pascaoperasi berikutnya. Teknik ini menggabungkan data MRI spesifik pasien dan atlas anatomi. Ketidakpastian dikodekan dan dievaluasi di permukaan menggunakan berbagai teknik seperti isoline atau enkode berbasis grid. Selanjutnya, teknik serupa diterapkan pada data permukaan biomolekuler [ SLK*22 , SMCL24 ]. Demikian pula, teknik yang sering digunakan untuk struktur biomolekuler terkadang juga diterapkan pada data medis. Uncertainty hull—salah satu bentuk visualisasi ketidakpastian paling awal untuk biomolekul [ RJ99 ]—juga telah diterapkan pada data medis [ GWHA18 ]. Sementara itu, beberapa teknik tidak memiliki padanan langsung dalam visualisasi biomolekuler, seperti metode animasi probabilistik yang dikembangkan untuk mengilustrasikan ketidakpastian dalam rendering volume medis [ LLPY07 ]. Metode ini menggunakan model fungsi transfer probabilistik untuk menyorot daerah yang tidak pasti, membantu dalam tugas-tugas seperti klasifikasi dan diagnosis jaringan. Mengingat penerapannya dalam visualisasi medis, mungkin berharga untuk menyelidiki potensinya untuk visualisasi biomolekuler.
5. Taksonomi
Berikutnya, kami menentukan cakupan dan proses pemilihan kami (Bagian 5.1) dan merinci skema klasifikasi kami (Bagian 5.2).
5.1. Ruang Lingkup
Kami fokus pada publikasi yang memperkenalkan atau mengadaptasi UAV di bidang visualisasi struktur biomolekuler. Pustaka daring yang kami telusuri adalah ACM Digital Library, IEEEXplore, Computers & Graphics (ScienceDirect) dan Eurographics Digital Library , serta PubMed . Penelusuran di masing-masing pustaka dilakukan menggunakan kombinasi istilah penelusuran:
- Biomolekul | Molekul | Protein
- Ketidakpastian | Ensemble | Fleksibilitas | Probabilitas | Kepadatan
- Visualisasi
Bagian pertama pencarian dimaksudkan untuk menemukan karya yang mempertimbangkan data molekuler. Pada bagian kedua, kami mencari pendekatan yang menangani ketidakpastian. Karena ketidakpastian bukanlah istilah yang unik dan memiliki banyak sinonim, kami juga mencari istilah terkait ensemble, fleksibilitas, probabilitas, dan densitas. Tujuan dari bagian terakhir adalah untuk menemukan pendekatan yang menyertakan komponen visual. Agar dapat dipertimbangkan dalam laporan kami, sebuah publikasi perlu menangani UAV data molekuler spasial atau sekuensial. Dengan batasan ini, kami mengecualikan pendekatan UV yang lebih umum seperti visualisasi grafik atau peta panas, di mana data biomolekuler hanya merupakan satu area aplikasi yang memungkinkan. Publikasi tersebut juga memerlukan setidaknya satu gambar UAV.
Meskipun istilah pencarian kami mencakup istilah terkait seperti fleksibilitas, publikasi perlu memvisualisasikan ketidakpastian (lihat Bagian 4). Misalnya, konsep seperti respons alosterik—perubahan bentuk atau aktivitas protein sebagai hasil pengikatan molekul di lokasi selain lokasi aktif—juga sering disebut sebagai fleksibilitas protein, tetapi visualisasi respons alosterik bukanlah visualisasi ketidakpastian dan dengan demikian tidak termasuk dalam survei kami. Selain metode pencarian utama kami, kami menggunakan strategi analisis kutipan rekursif. Lebih khusus lagi, kami mengidentifikasi makalah relevan yang dirujuk dalam sumber yang telah kami tinjau. Dengan menelusuri kutipan ke belakang, kami bertujuan untuk mengungkap publikasi tambahan yang relevan tetapi sebelumnya tidak dipertimbangkan. Secara total, kami mengidentifikasi 71 publikasi yang memenuhi kriteria seleksi kami, termasuk 10 karya tindak lanjut, yang menghasilkan 61 proyek berbeda.
5.2. Skema Klasifikasi
Skema klasifikasi kami terdiri dari tiga dimensi utama: jenis molekul, manifestasi ketidakpastian, dan strategi pemetaan, lihat Gambar 1. Untuk dimensi jenis molekul, kami mengkategorikan molekul menjadi molekul kecil Gambar, protein Gambar, dan asam nukleat Gambar. Di Bagian 3.1, kami membahas karbohidrat dan lipid sebagai jenis molekul tambahan yang berbeda. Sementara skema kami dapat diperluas untuk menyertakan kategori ini, sejauh pengetahuan kami, tidak ada penelitian tentang UAV yang khusus untuk jenis molekul ini. Kami membahas alasan kesenjangan ini di Bagian 9. Dalam visualisasi tempat beberapa molekul berinteraksi, kami mengklasifikasikan visualisasi berdasarkan jenis molekul primer—molekul yang menjadi fokus visualisasi.
Ada banyak manifestasi ketidakpastian yang potensial dalam data biomolekuler. Untuk laporan ini, kami mengklasifikasikan manifestasi ketidakpastian ke dalam lima kategori utama: ketidakpastian posisi Gambar, ketidakpastian dalam struktur sekunder Gambar, ketidakpastian dalam sifat fisikokimia Gambar, ketidakpastian mengenai molekul yang berinteraksi Gambar, dan ketidakpastian mengenai rongga biomolekuler Gambar. Kami membahas sumber ketidakpastian posisi di Bagian 4. Banyak sumber data struktur biomolekuler, seperti berkas struktural dari PDB [ BWF*00 ], menyertakan data ketidakpastian posisi seperti faktor-b atau serangkaian kemungkinan konformasi molekuler. Akibatnya, ketidakpastian posisi umumnya mudah diakses, dan banyak visualisasi ketidakpastian berfokus pada manifestasi ini. Seperti dibahas di Bagian 4, ketidakpastian posisi dapat menyebar melalui pemodelan dan komputasi berikutnya, sering kali bergabung dengan jenis ketidakpastian lain untuk membentuk manifestasi yang lebih kompleks. Meskipun beberapa manifestasi—seperti ketidakpastian dalam struktur sekunder—pada akhirnya berasal dari ketidakpastian posisi, penting untuk membedakannya. Hanya menyajikan data ketidakpastian posisi mentah tidak cukup menjawab banyak pertanyaan spesifik yang terkait dengan struktur sekunder. Mengingat perlunya pembedaan, kami telah mengidentifikasi kategori kami untuk menangkap berbagai aspek ketidakpastian dalam struktur biomolekuler. Kami memilih kategori utama dengan sifat-sifat yang penting bagi penelitian UV yang khususnya relevan bagi para ilmuwan yang mempelajari struktur biomolekuler. Kategori yang dipilih tidak menyeluruh tetapi menawarkan kerangka kerja praktis untuk memahami dan menganalisis ketidakpastian. Kategori tambahan dapat diperkenalkan jika diperlukan dalam penelitian mendatang. Tidak semua jenis ketidakpastian berlaku secara universal di seluruh jenis biomolekul. Ketidakpastian struktur sekunder relevan dengan protein dan asam nukleat, karena biomolekul yang lebih besar ini membentuk elemen atau motif struktural yang berulang. Molekul kecil terlalu kecil dan padat untuk menunjukkan struktur sekunder. Rongga terutama relevan dengan protein, di mana rongga sangat penting untuk fungsi seperti pengikatan ligan. Sementara asam nukleat dapat membentuk rongga dalam konteks tertentu, rongga tersebut kurang umum dan kurang dipelajari dibandingkan dengan protein. Molekul kecil umumnya terlalu kecil untuk membentuk rongga yang tepat, meskipun rongga tersebut mungkin menunjukkan lekukan.
Dimensi ketiga skema klasifikasi kami, strategi pemetaan, mengkategorikan pendekatan terutama ke dalam dua kelompok: memvisualisasikan distribusi ketidakpastian dan memvisualisasikan statistik ringkasan. Untuk perincian yang lebih rinci, kami membagi visualisasi distribusi ke dalam empat kategori: tampilan eksplisit Gambardan implisit , animasi , dan visualisasi jalur. Statistik ringkasan dapat dibagi lagi menjadi tampilan rentang , glif , dan atribut visual ukuran , warna , dan kepadatan . Kategori yang lebih rinci ini tidak lengkap dan dapat berkembang seiring dengan kemajuan penelitian dalam visualisasi struktur biomolekuler. Kami terutama mengantisipasi atribut visual tambahan seiring dengan kemajuan bidang ini.GambarGambarGambarGambarGambarGambarGambarGambar
Selain dimensi klasifikasi utama ini, kami mendokumentasikan tugas-tugas umum yang didukung oleh visualisasi, ketersediaan implementasi metode, dan strategi evaluasi metode yang diusulkan. Kami mendefinisikan empat jenis tugas abstrak untuk menggeneralisasi tujuan visualisasi ini yang seringkali sangat spesifik dan membantu pembaca mengidentifikasi metode yang sesuai. GambarTugas analisis mencakup kasus-kasus di mana detail data ketidakpastian perlu terlihat jelas. Ini mungkin melibatkan identifikasi wilayah-wilayah di mana ketidakpastian berada dalam rentang nilai tertentu atau membandingkan nilai-nilai ketidakpastian pada posisi-posisi yang berbeda dalam suatu molekul. GambarTugas perbandingan berfokus pada membandingkan molekul-molekul yang berbeda, secara eksplisit mengecualikan visualisasi ansambel dari beberapa konformasi molekuler. GambarTugas integrasi berlaku ketika ketidakpastian perlu dipertimbangkan bersama dengan informasi lainnya. Misalnya, ini dapat berarti memeriksa potensi elektrostatik dan ketidakpastian posisi secara bersamaan, yang mengharuskan kedua properti tersebut ditampilkan bersama-sama untuk memfasilitasi penilaian efek gabungannya. Terakhir, Gambartugas ikhtisar memungkinkan pemahaman yang luas tentang ketidakpastian secara sekilas, memastikan bahwa molekul dan ketidakpastian terkaitnya terlihat dalam bentuk statis tanpa interaksi pengguna.
Untuk membantu pembaca dalam memilih UAV yang tepat, kami menetapkan persyaratan yang jelas: suatu metode visualisasi memenuhi syarat untuk atribut tugas tertentu hanya jika UAV molekul tersebut secara langsung mendukung tugas tersebut. Visualisasi tambahan—seperti diagram panas dalam kerangka kerja yang lebih besar—tidaklah cukup. Selain itu, suatu metode harus menyajikan contoh visualisasi yang menunjukkan tugas tersebut atau menjelaskan bagaimana metode tersebut mendukungnya.
Dalam bab-bab berikut, kami mengulas publikasi yang membahas UV molekuler. Tabel 1 mencantumkan semua metode yang dipertimbangkan dan mengklasifikasikannya menurut skema kami.
 |
6. Molekul KecilGambar
Meskipun metode yang dibahas dalam bagian ini diilustrasikan dengan molekul kecil, metode tersebut tidak terbatas pada molekul kecil. Teknik tertentu, khususnya yang mengandalkan representasi bola dan tongkat, mungkin tidak optimal untuk molekul yang lebih besar, meskipun secara teoritis, metode tersebut masih dapat digunakan. Di sisi lain, metode yang menggunakan visualisasi volume seharusnya lebih mudah diterapkan pada berbagai jenis biomolekul.
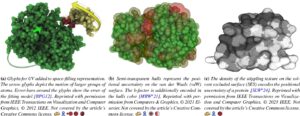
Eksplisit Gambar Pada tahun 1999, Rheingans dan Joshi adalah orang pertama yang menulis publikasi secara eksplisit tentang UV molekuler [ RJ99 ]. Mereka mengusulkan tiga opsi untuk memvisualisasikan molekul dengan ketidakpastian posisi (Gambar 2 ). Dua metode didasarkan pada volume kemungkinan yang dihitung dari ansambel. Volume kemungkinan menyimpan probabilitas sebuah atom berada di setiap voksel. Mereka memberikan dua opsi untuk memvisualisasikan volume ini. Visualisasi volume pertama mengekstrak isosurfaces transparan dari volume dan merendernya di atas visualisasi bola-dan-tongkat yang buram (Gambar 2b ). Metode visualisasi volume lainnya merender volume secara langsung, menghasilkan tampilan seperti awan (Gambar 2c ). Metode ketiga menumpangkan setiap konformer dalam ansambel menggunakan representasi bola-dan-tongkatnya. Setiap konformasi dapat dirender secara buram, atau semi-transparan (Gambar 2a ) sehingga daerah yang lebih pasti tampak buram sementara daerah yang tidak pasti tampak lebih transparan.
Schmidt-Ehrenberg dkk. [ SBH02 ] menggunakan direct volume rendering (DVR) atau isosurfaces buram untuk menampilkan volume kemungkinan. Mereka berfokus pada konformasi metastabil, yang merupakan keadaan di mana molekul bertahan untuk waktu yang lama. Oleh karena itu, mereka memvisualisasikan beberapa konformasi metastabil dari suatu molekul sekaligus, sementara juga menampilkan volume kemungkinan untuk setiap konformasi metastabil secara terpisah.
Implicit Gambar Best dan Hege [ BH02 ] memungkinkan pengguna untuk mengelompokkan atom menjadi ellipsoid dalam aplikasi mereka untuk mengurangi kekacauan dan dengan demikian memudahkan pendeteksian perubahan konformasi. Selain itu, mereka memadukan beberapa kerangka waktu simulasi molekuler untuk memfasilitasi pendeteksian perubahan besar. Mirip dengan pemetaan implisit yang diusulkan oleh Rheingans dan Joshi [ RJ99 ], Sabando et al. [ SUS*21 ] memvisualisasikan kesamaan senyawa kimia dengan melapisi visualisasi semi-transparan dari konformasi individual. Mereka menambahkan opsi untuk membalikkan opasitas. Hasilnya, bagian molekul yang lebih tidak pasti dapat disorot dengan tampak lebih buram.
Rentang Gambar Knoll et al. [ KCL*13 ] mendefinisikan interval antara jari-jari ikatan kimia dan jari-jari vdW sebagai interval ketidakpastian atas distribusi kerapatan muatan. Mereka membuat molekul dalam warna hitam menggunakan representasi bola dan tongkat. Kemudian, mereka melapisi visualisasi volume kerapatan elektron. Mereka menggunakan DVR dengan fungsi transfer khusus. Dalam sebagian besar contoh mereka, mereka menggunakan dua puncak untuk menampilkan isolevel ikatan kimia dalam warna biru dan isolevel yang sesuai dengan jari-jari vdW dalam warna merah. Dalam makalah mereka, mereka berfokus pada antarmuka molekuler daripada biomolekul. Karena menggunakan representasi bola dan tongkat, visualisasi mereka cocok untuk molekul kecil daripada biomolekul yang lebih besar.
Ukuran Gambar Wagner dan Himmel [ WH17 ] menggunakan warna dan ukuran untuk mengkodekan RMSD antara dua struktur molekul dalam representasi bola dan tongkat yang dimodifikasi. Warna bola menunjukkan RMSD absolut pasangan atom. Ukuran bola sebanding dengan kontribusi relatif pasangan atom terhadap RMSD total.
Metode yang dikembangkan untuk molekul kecil umumnya memberikan gambaran visual ketidakpastian. Metode ini menunjukkan keberadaan ketidakpastian dalam molekul dan memberikan gambaran kasar tentang besarnya ketidakpastian. Namun, selain kode warna dan ukuran gabungan oleh Wagner dan Himmel [ WH17 ], membuat penilaian yang lebih rinci mungkin sulit. Metode ini juga tidak memfasilitasi tugas perbandingan atau integrasi.
7. ProteinGambar
Protein merupakan biomolekul yang paling banyak dipelajari untuk UV, dengan visualisasi yang tersedia untuk semua jenis manifestasi ketidakpastian dan pemetaan sesuai dengan skema kami. Mirip dengan molekul kecil, banyak metode untuk UV berlaku untuk jenis biomolekul lainnya. Meskipun demikian, teknik tertentu bersifat khusus untuk protein, seperti visualisasi struktur sekunder atau rongga.
7.1. PosisiGambar
Eksplisit Gambar Sejak awal tahun 1989, Goodsell et al. [ GMO89 ] mulai memvisualisasikan distribusi molekul daripada posisi atom yang tepat. Mereka menggunakan rendering volume dengan berbagai fungsi transfer untuk memvisualisasikan data molekuler volumetrik, termasuk kerapatan elektron dan potensial elektrostatik. Secara khusus, mereka mengusulkan tiga fungsi transfer. Satu menghasilkan representasi awan, dan dua lainnya menghasilkan representasi permukaan. Representasi awan menetapkan opasitas yang lebih tinggi ke kepadatan yang lebih tinggi, menghasilkan area tak pasti yang lebih transparan. Salah satu representasi permukaan menggabungkan beberapa isosurface buram dengan lapisan yang berbeda menjadi terlihat saat menggunakan bidang kliping. Representasi lainnya terdiri dari isosurface transparan bertumpuk yang menjadi lebih buram saat kepadatan dalam volume meningkat. Lee dan Varshney [ LV02 ] mengusulkan metode isosurface lain: permukaan molekul fuzzy, versi SES yang sadar ketidakpastian. Mereka merender beberapa permukaan semi-transparan bertumpuk untuk membuat permukaan fuzzy ini. Berbeda dengan kebanyakan metode lain, Lee dan Varshney tidak menghitung permukaan mereka dari data volume. Mengingat data koordinat molekul, mereka menghitung bola p-probabilitas untuk setiap atom. Bola p-probabilitas ini adalah bola terkecil yang berisi pusat atom dengan probabilitas p . Mereka memilih beberapa tingkat p untuk merender permukaan fuzzy. Untuk setiap p , mereka memperluas semua bola p-probabilitas dengan radius probe. Kemudian, untuk setiap p , mereka menghitung dan merender permukaan gaya SES semi-transparan. Permukaan molekul fuzzy tampak buram dibandingkan dengan permukaan molekul tanpa representasi ketidakpastian. Krone et al. [ KBE09 ] juga menggunakan cangkang bersarang dari permukaan molekul semi-transparan untuk menunjukkan ketidakpastian posisi protein (Gambar 6 ). Mereka menghitung kerapatan probabilitas spasial dari data simulasi pada grid reguler. Selanjutnya, mereka menghasilkan dan merender permukaan yang sesuai dengan rentang probabilitas yang berbeda dengan menempatkan bola atom virtual dalam grid probabilitas dan menghitung SES untuk setiap rentang. Lapisan dengan ketidakpastian yang lebih tinggi dirender lebih transparan daripada yang dengan ketidakpastian yang lebih rendah. [ HCZP06 ] menggunakan DVR untuk memvisualisasikan data volume 3D yang dihasilkan oleh simulasi mekanika kuantum protein. Punjani dan Fleet [ PF23] mengembangkan 3DFlex, jaringan saraf berbasis gerakan untuk menentukan struktur dan gerakan protein dari data cryo-EM. Variabel laten model menggambarkan kemungkinan deformasi molekul. Mereka memvisualisasikan beberapa isosurfaces sepanjang satu dimensi laten yang berkisar dari minus hingga plus satu deviasi standar.
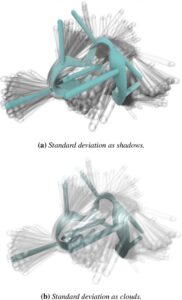
Implisit Gambar Visualisasi ensemble sangat erat kaitannya dengan visualisasi distribusi secara langsung. Dalam representasi implisit ini, konformasi molekuler individual ditumpangkan untuk merepresentasikan distribusi kontinu. Pada tahun 1997, Kelley dan Sutcliffe. [ KS97 ] mengusulkan basis data daring OLDERADO yang memfasilitasi tampilan ansambel konformasi molekuler dan konformasi paling representatif dalam jendela terpisah. Konformasi ditampilkan menggunakan representasi tongkat. Lai dkk. [ LKCW04 ] memvisualisasikan ansambel menggunakan tongkat, pita, atau representasi kartun. Server web MOBI [ MWT10 ] memvisualisasikan ansambel NMR protein dan mengodekan mobilitas residu dalam warna. Heinrich dkk. [ HKOW14 ] menyajikan sistem visualisasi untuk memvisualisasikan daerah yang tidak teratur secara intrinsik (IDR). Mereka menggabungkan koordinat paralel dengan visualisasi ensemble. Penyikatan dan penautan antara koordinat paralel dan ansambel memungkinkan pemeriksaan dan analisis protein yang lebih baik. Melvin dan Salsbury [ MS16 ] secara khusus memilih konformasi yang akan ditumpangkan untuk menciptakan visualisasi ensembel yang lebih mudah dikelola. Salah satu kriteria pemilihan yang mereka usulkan adalah bingkai yang terletak dalam satu deviasi standar dari median. Untuk meminimalkan kekacauan visual, mereka menggunakan teknik fokus dan konteks: Mereka menampilkan satu konformasi representatif yang buram, yang lain transparan, sehingga menghasilkan visualisasi seperti awan atau bayangan (Gambar 7 ).
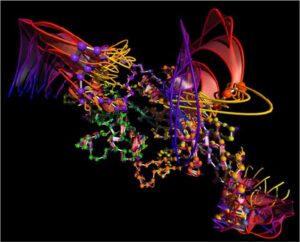
Pathlines Gambar Dabdoub et al. [ DMR08 , DRSR15 ] menggambar pathlines yang menelusuri pergerakan atom-atom individual dalam molekul dari waktu ke waktu (Gambar 8 ). Studi pendahuluan mereka menunjukkan bahwa visualisasi pathline dapat membantu memahami gerakan molekuler. Namun, contoh-contoh mereka difokuskan pada molekul yang relatif kecil. Menggunakan teknik ini untuk molekul yang lebih besar atau dalam banyak kerangka waktu dapat menyebabkan tampilan yang berantakan.
Animasi Gambar Fisher et al. [ FTPG90 ] memvisualisasikan ketidakpastian posisi dengan merender permukaan konsensus dari dua permukaan protein yang dipindahkan secara maksimal. Warna permukaan bervariasi dari waktu ke waktu, sesuai dengan perubahan medan elektrostatik. Sebagai opsi visualisasi kedua, potensial elektrostatik diberi kode warna pada permukaan protein, dan medan elektrostatik ditunjukkan dengan anak panah. Dengan metode ini, animasi membuat perubahan terlihat dari waktu ke waktu. Metode ini secara inheren mengintegrasikan beberapa jenis data dan memungkinkan beberapa tingkat analisis. Namun, ini adalah satu-satunya metode dalam literatur yang dikumpulkan yang tidak menawarkan ikhtisar sekilas.
Ukuran Gambar Berbeda dengan pemetaan rentang, yang menunjukkan parameter distribusi, metode yang menggunakan pemetaan ukuran kurang dibatasi. Daripada memetakan atribut distribusi secara langsung, metode berbasis ukuran dapat, misalnya, menggunakan ukuran untuk menyampaikan bahwa nilai yang lebih besar sesuai dengan ketidakpastian yang lebih tinggi. Koradi dkk. [ KBW96 ] mengembangkan perangkat lunak visualisasi MOLMOL. Ketidakpastian posisi dapat ditampilkan oleh superposisi beberapa konformer atau dengan memvariasikan radius tabung dari presentasi tulang punggung. Tabung yang lebih lebar memiliki ketidakpastian posisi yang lebih tinggi. Pengodean ukuran khusus ini sering disebut representasi sosis atau dempul. Contoh representasi sosis adalah penggambaran tulang punggung protein pada Gambar 9b . Scott dan Straus [ SS15 ] menggunakan visualisasi ansambel dan metafora sosis dalam visualisasi gabungan. Karena saluran warna tetap tersedia untuk mengodekan properti yang berbeda, teknik ini mendukung tugas integrasi di samping tugas ikhtisar. Kesesuaiannya untuk tugas analisis atau perbandingan tidak jelas, karena tidak ada evaluasi atau contoh aplikasi yang menunjukkan keefektifannya dalam konteks ini.
Warna Gambar Pilihan yang sangat populer untuk menampilkan statistik ringkasan adalah penggunaan warna. Tidak ada pemetaan warna standar untuk ketidakpastian. Yang relatif umum adalah peta warna biru-putih-merah. Namun, peta warna ini juga sangat terkait dengan potensial elektrostatik. Pilihan populer lainnya, peta warna merah-hijau, bermasalah bagi orang dengan kekurangan penglihatan warna. Beberapa metode yang dibahas sebelumnya menggunakan warna sebagai metode pemetaan tambahan. Di sini, kami membahas metode yang terutama menggunakan atribut warna. Pada tahun 1995, Zachmann et al. [ ZKB95 ] menggunakan pemetaan tekstur untuk memetakan fleksibilitas molekuler ke SES. Thorpe et al. [ TLR*01 ] menggunakan teori kendala untuk menghitung fleksibilitas protein dan mewarnai protein mereka sesuai dengan ukuran fleksibilitas ini. Falk et al. [ FTB*24 ] mengusulkan kerangka kerja visual untuk mendukung pembangunan model dari peta kepadatan cryo-EM. Ini memfasilitasi verifikasi dan validasi model atom 3D yang dibangun dari peta ini. Beberapa ukuran kualitas ditampilkan sebagai peta panas, dan untuk analisis spasial, ukuran yang dipilih pengguna dapat dipetakan ke representasi 3D model. Sementara pemetaan visual data ketidakpastian relatif sederhana, verifikasi model 3D secara tradisional bergantung pada tampilan tabular atau grafik garis sederhana. Pendekatan ini merupakan langkah maju yang penting dalam mendukung pembangunan model.
Pengodean warna menawarkan keuntungan dalam menyediakan penggambaran ketidakpastian yang terperinci, sehingga secara umum sangat cocok untuk tugas analisis dan perbandingan. Meskipun kedua metode yang disebutkan secara khusus dalam bagian ini tidak secara eksplisit menunjukkan contoh perbandingan, tampilan berdampingan biasanya layak untuk tugas tersebut [ MRW*21 ]. Selain itu, warna membantu menyediakan ikhtisar data. Namun, karena warna mengodekan informasi ketidakpastian, warna tidak dapat mewakili atribut lainnya. Akibatnya, metode tersebut tidak cocok untuk tugas integrasi.
Kepadatan Gambar Mirip dengan pemetaan warna, tekstur yang berbeda dapat mengodekan ketidakpastian. Satu keuntungan menggunakan tekstur yang tidak berbasis warna adalah bahwa tekstur tersebut dapat dikombinasikan dengan warna untuk merepresentasikan beberapa atribut. Misalnya, warna dapat digunakan untuk atribut primer seperti potensial elektrostatik, sementara tekstur dapat merepresentasikan atribut sekunder seperti ketidakpastian. Sterzik dkk. [ SMCL24 ] mengevaluasi persepsi tiga peta tekstur ilustratif (stippling, hatching, dan triangles) dengan kepadatan primitif yang bervariasi (Gambar 4c ). Mereka melakukan evaluasi persepsi crowdsourced dan memberikan reparameterisasi seragam persepsi yang kira-kira untuk nilai kepadatan. Sterzik dkk. [ SLK*22 , SLK*23 ] mengusulkan penggunaan garis fitur bergaya untuk mengodekan ketidakpastian dalam visualisasi biomolekuler (Gambar 10 ). Mereka memodifikasi atribut garis blur, sketchiness, dashing, width, dan grayscale (nilai warna) untuk merepresentasikan berbagai tingkat ketidakpastian. Selain itu, mereka melakukan dua studi crowdsourced untuk mengevaluasi efektivitas variabel garis ini dalam membedakan nilai ketidakpastian. Di antara atribut yang diuji, mereka mengidentifikasi lebar garis sebagai yang paling sesuai untuk membedakan secara efektif antara berbagai tingkat ketidakpastian.

Glif Glif Gambar adalah metafora pemetaan lain yang kurang umum. Bryden et al. [ BPG12 ] menggunakan glif panah untuk menampilkan gerakan yang lebih besar dari kelompok molekuler. Mereka memvisualisasikan hasil analisis mode normal untuk menggambarkan fleksibilitas molekuler dengan meringkas kelompok atom menjadi kelompok yang lebih besar dan memvisualisasikan gerakan kelompok dengan glif panah. Meskipun gerakan yang lebih besar ini mungkin tidak secara ketat mewakili ketidakpastian, atribut glif dapat mengodekan kesalahan model pemasangan dan kekakuan kelompok. Properti ini membantu dalam menilai keandalan model gerakan. Mereka memvisualisasikan kesalahan dalam model pemasangan mereka dengan garis-garis kesalahan di sekitar glif. Tingkat kesalahan didiskritisasi menjadi tiga tingkat: kesalahan kecil, sedang, dan kesalahan yang lebih besar. Pengodean masing-masing adalah tidak ada garis kesalahan, garis kesalahan kecil, dan garis kesalahan besar. Selain itu, mereka menambahkan pola garis pada anak panah untuk mengodekan kekakuan kelompok molekuler. Pola garis juga memiliki tiga tingkat diskrit: berwarna lengkap, garis-garis padat, dan garis-garis jarang, dari energi nonrigid rendah ke tinggi (Gambar 4a ). Bedoucha et al. [ BRHB20 ] memasukkan glif ini ke dalam alat analisis visual mereka untuk analisis mode normal. Selain itu, fluktuasi atom ditunjukkan dengan pemetaan warna. Selain mendukung tugas ikhtisar dan analisis, glif memungkinkan integrasi data tambahan dengan menambahkan elemen visual terpisah ke representasi. Namun, karena ditempatkan pada posisi diskrit—dan dalam metode yang dibahas di sini, agak jarang—glif hanya dapat menyampaikan informasi terperinci pada titik-titik tertentu. Hal ini membuatnya tidak cocok untuk tugas yang memerlukan representasi ketidakpastian yang berkelanjutan di semua posisi.
7.2. Struktur SekunderGambar
Tiga makalah Implicit Gambar menggunakan ensemble untuk membandingkan struktur sekunder. Kocincová et al. [ KJB*17 ] memperkenalkan sistem untuk membandingkan struktur sekunder ensemble protein. Sistem ini mengintegrasikan tampilan ensemble 3D tradisional dengan dua tampilan yang secara langsung membandingkan elemen struktur sekunder. Tampilan perbandingan terdiri dari urutan yang ditumpangkan dan disandingkan. Elemen struktur sekunder digambarkan menggunakan simbol konvensional: panah untuk lembar beta, spiral untuk heliks alfa, dan garis untuk kumparan. Meskipun tampilan yang ditumpangkan dan disandingkan disajikan dalam format 2D yang diratakan, mereka mempertahankan dan mengodekan beberapa informasi tentang orientasi timbal balik elemen yang sesuai melalui sudut relatif terhadap garis horizontal. Chen dan Porter [ CP23 ] juga menampilkan penyelarasan urutan protein yang disandingkan dengan simbol yang mengodekan elemen struktur sekunder. Urutan diwarnai dengan skor konservasi, faktor-b, atau ukuran yang ditentukan pengguna.
Kedua metode secara eksplisit mendukung perbandingan. Pendekatan oleh Kocincová et al. [ KJB*17 ] mempertahankan beberapa informasi orientasi dalam tampilan 2D, sedangkan Chen dan Porter [ CP23 ] meratakan urutan secara menyeluruh. Namun, kode warna Chen dan Porter memungkinkan penilaian kuantitatif dari ukuran ketidakpastian skalar.
Schulz dkk. [ SSK*18 ] memanfaatkan sekuens yang disandingkan untuk membandingkan penugasan struktur sekunder yang dihasilkan oleh metode yang berbeda. Jika sekuens konsensus tersedia, sekuens tersebut ditampilkan sebagai sekuens tunggal. Dalam kasus ketidakpastian penugasan, beberapa sekuens ditumpuk satu sama lain. Untuk lebih menggambarkan ketidakpastian, mereka memperkenalkan kombinasi distorsi geometrik dan transparansi dalam representasi kartun mereka. Pita dan tabung yang digunakan untuk menggambarkan elemen struktur sekunder didistorsi menggunakan fungsi sinus atau segitiga, dengan frekuensi dan amplitudo yang mencerminkan tingkat ketidakpastian (Gambar 5 ). Selain itu, prediksi struktural yang berbeda (misalnya, heliks, lembaran, atau kumparan) dapat dilapiskan menggunakan transparansi pintu kasa, di mana struktur yang lebih mungkin tampak lebih buram. Mereka juga menunjukkan penerapan teknik distorsi mereka untuk menggambarkan ketidakpastian posisi dalam representasi tabung tulang punggung. Alat mereka adalah satu-satunya yang ada dalam literatur yang dikumpulkan untuk membandingkan berbagai metode penugasan struktur sekunder, tetapi mereka juga menunjukkan kasus penggunaan untuk jenis ketidakpastian lainnya, khususnya ketidakpastian posisi
Warna Gambar Alat Aquaria [ OSK*15 ] menggunakan pemodelan homologi, sebuah metode yang memprediksi struktur 3D protein berdasarkan kemiripannya dengan struktur yang diketahui (templat). Alat ini menghasilkan model struktural dengan menyelaraskan urutan input dengan templat ini, dengan asumsi bahwa urutan yang serupa akan mengadopsi struktur yang serupa. Model ini diwarnai berdasarkan dua atribut: Elemen struktur sekunder menentukan rona, sementara tingkat percakapan antara yang paling relevan dan struktur input ditunjukkan dengan meredam warna. Daerah yang sangat kekal diwarnai cerah, daerah yang tidak kekal digambar dalam warna hitam, dan bagian yang disisipkan diwarnai abu-abu. Semua struktur terkait lainnya ditampilkan dalam tampilan 2D sebagai urutan persegi panjang, diwarnai menurut skema yang disebutkan di atas, memberikan gambaran umum dari semua urutan. Heinrich dkk. [ HKO15 ] mengevaluasi pemetaan warna ini dalam studi crowdsourced. Mereka menemukan korelasi yang signifikan antara kualitas gambar yang dipersepsikan dan kualitas penyelarasan.
Vázquez et al. [ VHG*18 ] mengembangkan alat analisis visual untuk interaksi protein-ligan yang merepresentasikan ketidakpastian struktur posisional dan sekunder. Protein digambarkan sebagai susunan melingkar dari residunya, dengan ligan secara opsional ditampilkan di bagian tengah. Ketidakpastian posisional dapat disertakan dengan menambahkan batang pada residu, di mana tinggi dan warna mengkodekan RMSF. Selain itu, ikatan hidrogen intra-molekul divisualisasikan sebagai pita yang menghubungkan residu masing-masing, dengan warna pita menunjukkan persistensi ikatan. Meskipun ini adalah visualisasi struktural yang sangat abstrak, kami menyertakan kasus tepi ini dalam survei kami karena kemiripannya dengan beberapa contoh UAV untuk RNA yang ada (lihat Bagian 8.1). Untuk mengurangi keterbatasan representasi abstrak tersebut, penulis menyertakan tampilan struktur 3D yang terpisah; namun, tampilan ini tidak menyertakan visualisasi ketidakpastian.
7.3. FisikokimiaGambar
Rasheed et al. [ RCBB16b , RCBB19 ] mengembangkan kerangka statistik untuk mengukur ketidakpastian untuk QOI yang ditentukan pengguna, seperti potensi Poisson-Boltzmann. Mereka mengusulkan memvisualisasikan ketidakpastian ini melalui DVR atau pemetaan warna. Lebih jauh, probabilitas pengikatan ligan divisualisasikan dengan memetakan warna ke permukaan protein dan ligan. Dalam pracetak [ RCBB16a ], mereka juga memperkenalkan metode untuk memvisualisasikan ketidakpastian posisi menggunakan glif vektor. Pada setiap posisi atom, tiga glif panah mewakili variabilitas lokasi atom, dengan panjang glif menunjukkan besarnya ketidakpastian. Sementara beberapa metode lain memperhitungkan sifat fisikokimia, ini adalah satu-satunya yang memiliki fokus yang jelas pada mereka. Visualisasi yang diusulkan mendukung tugas analisis, perbandingan, dan ikhtisar.
7.4. Rongga gigiGambar
Sementara metode yang dibahas sebelumnya memvisualisasikan manifestasi ketidakpastian yang menyangkut keseluruhan molekul, paragraf berikut membahas metode yang dikembangkan untuk menampilkan ketidakpastian untuk rongga biomolekuler.
Color Gambar Ashford et al. [ AMA*12 ] memvisualisasikan ketidakpastian mengenai kantong molekuler. Probabilitas atom atau residu yang termasuk dalam kantong dipetakan warnanya ke permukaan, sehingga mendukung tugas analisis dan tinjauan umum.
Implicit Gambar Lindow et al. [ LBBH12 ] mengusulkan beberapa visualisasi untuk dinamika rongga dan terowongan protein. Mereka menghitung probabilitas tempat tinggal untuk setiap titik pada grid reguler dan memvisualisasikan probabilitas ini sebagai iso-surfaces. Probabilitas tempat tinggal didefinisikan sebagai “proporsi waktu di mana titik berada di dalam rongga”. Selain itu, mereka menggunakan dua jenis visualisasi ensemble 2D. Grafik garis menunjukkan perubahan topologi dalam saluran dan rongga. Setiap komponen—jaringan jalur melalui molekul yang mendefinisikan rongga atau saluran tunggal—ditampilkan sebagai polyline tunggal. Jika pemisahan atau penggabungan komponen terjadi selama simulasi, garis-garis tersebut terbagi atau bergabung masing-masing. Grafik garis kedua menunjukkan perluasan komponen dari waktu ke waktu. Dalam grafik ini, posisi dan lebar garis mewakili lokasi dan luas komponen jalur sepanjang arah yang ditentukan pengguna. Karena garis-garis dalam grafik ini dapat tumpang tindih, garis-garis tersebut digambar semi-transparan dan dicampur. Sementara visualisasi ketidakpastian spasial hanya memberikan gambaran umum data, alat tambahan dapat digunakan untuk tugas analisis.
Byška et al. [ BJG*15 ] memberikan visualisasi ikhtisar ketergantungan waktu dari kemacetan terowongan. Bentuk kemacetan divisualisasikan sebagai kontur, dan beberapa langkah waktu ditumpangkan untuk menghasilkan visualisasi ansambel. Kontur diwarnai sesuai dengan langkah waktu terjadinya. Kontur juga menunjukkan asam amino di sekitarnya dengan batang yang disusun secara radial di sekitar kontur berdasarkan posisinya. Warna batang menunjukkan sifat fisiko-kimia asam amino. Karena asam amino biasanya digantikan oleh asam amino lain seiring waktu, batang individual untuk setiap asam amino ditempatkan di samping satu sama lain. Tinggi batang menunjukkan jumlah langkah waktu asam amino menjadi bagian dari batas terowongan. Kemacetan disejajarkan sedemikian rupa sehingga asam amino di sekitarnya paling banyak tumpang tindih. AnimoAminoMiner [ BLMG*16 ], sebuah karya lanjutan, memvisualisasikan lebar terowongan di sepanjang panjangnya, menyajikan beberapa langkah waktu sebagai ansambel. Tampilan kedua memungkinkan eksplorasi asam amino di sekitar terowongan. Meskipun kedua metode oleh Byška dkk. memberikan representasi struktural yang sangat abstrak, kami memasukkannya dalam tinjauan ini karena terbatasnya jumlah karya tentang visualisasi ketidakpastian untuk terowongan.
Kedua metode tersebut terintegrasi ke dalam alat analisis Caver Analyst [ JBB*18 ]. Metode pertama oleh Byška et al. khususnya berguna ketika fokus utamanya adalah pada kemacetan terowongan. Jika tidak, ketergantungan waktu dari keseluruhan terowongan dapat diperiksa menggunakan AnimoAminoMiner [ BLMG*16 ] atau metode oleh Lindow et al. [ LBBH12 ]. Ketiga metode tersebut menggunakan representasi struktural 2D yang abstrak; namun, pendekatan oleh Lindow et al. [ LBBH12 ] juga menggabungkan visualisasi 3D.
7.5. Molekul yang berinteraksiGambar
Selanjutnya, kami membahas metode yang menggabungkan ukuran ketidakpastian untuk molekul yang berinteraksi dengan protein yang divisualisasikan. Sebagian besar metode yang dijelaskan di bawah ini berfokus pada distribusi molekul yang lebih kecil di sekitar molekul utama.
Warna Gambar Alharbi et al. [ AKCL18 , AKCL19a , AKCL19b ] memvisualisasikan interaksi protein-protein dan protein-lipid. Mereka menggabungkan jumlah kontak dengan protein lain atau lipid selama simulasi untuk setiap protein dan menunjukkan hasilnya menggunakan kode warna pada representasi abstrak protein (silinder). Demikian pula, Duncan et al. [ DCS20 ] memetakan frekuensi kontak lipid yang berbeda ke permukaan protein membran menggunakan warna. Kerangka kerja ProLint [ ST21 ] menggunakan teknik yang serupa tetapi memungkinkan pemilihan metrik yang berbeda untuk distribusi, seperti total kontak atau kontak terpanjang. Selain itu, ProLint memungkinkan visualisasi distribusi lipid melalui isolevel. Schatz et al. [ SFS*21 ] menghitung probabilitas kontak untuk ligan di seluruh permukaan molekul dengan menggabungkan seberapa sering ligan jenis tertentu bersentuhan dengan titik pada permukaan molekul. Kode warna menunjukkan kemungkinan titik kontak. Selain itu, mereka menyajikan Compressed Ligand Interaction Sequence Diagram, yang menunjukkan probabilitas kontak, RMSF, dan nilai-nilai lain per residu. Krone et al. [ KFS*17 ] juga menggunakan kode warna permukaan untuk menunjukkan ketidakpastian. Mereka memperkenalkan Molecular Surface Maps: proyeksi peta permukaan molekuler yang memberikan gambaran umum bebas oklusi dari titik-titik kontak berwarna pada permukaan. Mereka menunjukkan pendekatan mereka untuk beberapa properti, termasuk faktor-b protein dan probabilitas kontak untuk air. Metode dalam bagian ini berlaku untuk skenario yang berbeda tetapi berbagi teknik dasar yang sama. Warna digunakan untuk memetakan nilai ketidakpastian skalar yang terkait dengan molekul yang berinteraksi ke permukaan molekuler. Ini memberikan gambaran umum data dan mendukung tugas-tugas analisis.
Rentang Gambar Dalam literatur yang dikumpulkan, hanya satu makalah yang secara khusus berfokus pada interaksi protein-protein. Furmanová et al. [ FJK*20 ] mengembangkan alat analisis visual untuk ansambel besar kompleks protein multi-tubuh. Alat ini menampilkan tampilan 3D di mana distribusi protein sekunder di sekitar protein primer direpresentasikan menggunakan lambung isolevel. Pendekatan ini membantu mengurangi kekacauan visual yang akan muncul dari tampilan langsung ansambel lengkap dari kemungkinan konfigurasi. Selain itu, kompleks protein individual dapat dieksplorasi dalam tampilan meledak, di mana frekuensi kontak dipetakan ke permukaan menggunakan warna. Di luar representasi struktural, alat ini mencakup tampilan non-struktural yang menyediakan pemetaan ketidakpastian lebih lanjut, seperti peta panas frekuensi interaksi.
Bhati dkk. [ BHP*23 ] menampilkan okupansi ligan menggunakan permukaan isovalue wireframe. Metode ini mirip dengan beberapa visualisasi distribusi eksplisit yang dibahas di bawah ini; namun, representasi wireframe terkadang dapat mengaburkan permukaan yang mendasarinya, dan hanya satu isolevel yang disediakan, sehingga menyulitkan penilaian terperinci terhadap distribusi ketidakpastian. Meskipun demikian, metode ini menawarkan gambaran awal tentang distribusi ligan.
Visualisasi distribusi Gambar Besançon et al. [ BGB*18 , BWR*20 , BGB*20 ] berfokus pada kelas karbohidrat tertentu: n-glikana. Mereka memetakan distribusi karbohidrat ke permukaan protein, baik secara langsung dengan menambahkan opasitas konformasi glikana individual atau dengan mengabstraksi dan menggambar garis kontur untuk distribusi. Kedua versi visualisasi dapat dilihat pada Gambar 9a . Metode ini berada di antara visualisasi skalar dan distribusi. Seperti pendekatan berbasis warna, metode ini memetakan ketidakpastian ke warna di permukaan. Pada saat yang sama, mirip dengan visualisasi distribusi, metode ini merepresentasikan distribusi ketidakpastian tanpa terlebih dahulu mengubahnya menjadi nilai skalar untuk setiap titik pada permukaan molekuler. Seperti kebanyakan visualisasi distribusi lainnya, metode ini sangat cocok untuk mendapatkan gambaran umum data, tetapi detail yang lebih halus mungkin tidak cukup terlihat untuk tujuan analisis.
Bidmon et al. [ BGB*08 ] menyajikan sebuah metode untuk mengekstrak dan memvisualisasikan jalur molekul pelarut yang paling mungkin di dekat rongga protein. Mereka pertama-tama mengumpulkan jalur semua molekul pelarut yang memasuki area minat yang ditentukan pengguna selama simulasi. Jalur yang serupa dikelompokkan, dan hasilnya ditampilkan sebagai tabung, dengan ketebalan tabung menunjukkan jumlah jalur yang dikelompokkan. Artinya, jalur dengan probabilitas yang lebih tinggi lebih menonjol daripada yang kurang mungkin. Tabung diberi kode warna untuk menunjukkan arah pergerakan, dan pengguna dapat memfilter kelompok dengan probabilitas rendah. Vad et al. [ VBJ*17 ] mengembangkan sebuah sistem untuk mengeksplorasi lintasan air untuk simulasi MD protein. Sistem mereka juga memvisualisasikan lintasan yang secara opsional dapat dihaluskan. Selain itu, distribusi molekul air dapat divisualisasikan secara eksplisit dengan isosurfaces densitas. Karena memvisualisasikan secara langsung semua lintasan molekul yang berinteraksi mengarah ke tampilan yang sangat berantakan, kedua metode [ BGB*08 , VBJ*17 ] bergantung pada representasi yang disederhanakan. Evaluasi lebih lanjut diperlukan untuk menentukan kekurangan dan manfaatnya. Durrieu et al. [ DLB08 ] secara eksplisit memvisualisasikan distribusi molekul air dalam kompleks protein melalui DVR. Jurčík et al. [ JFB*19 ] mengembangkan alat analisis visual untuk analisis lintasan ligan untuk simulasi MD. Alat ini menawarkan tampilan 3D dengan dua penyandian ketidakpastian (Gambar 9b ). Pertama, mereka memvisualisasikan distribusi lintasan ligan berdasarkan kisi voxel. Kisi voxel dihitung dengan menghitung jumlah lintasan untuk setiap voxel. Setiap voxel divisualisasikan oleh bola yang ukuran dan warnanya sesuai dengan nilai kepadatan lokal. Kedua, fleksibilitas protein dikodekan dalam lebar tulang punggung (plot sosis). Skånberg et al. [ SFL*21 ] mengembangkan visualisasi serupa untuk mengeksplorasi hubungan spasial antara struktur atom. Distribusi molekul kecil ditentukan dengan menyediakan kerangka acuan internal untuk agregasi hasil yang bermakna. Untuk tujuan ini, struktur acuan (molekul besar) diasumsikan sebagai objek kaku. Molekul kecil diagregasi dari waktu ke waktu dan kategori molekuler. Metode ini mencakup dua opsi untuk menampilkan distribusi molekul kecil. Distribusi dapat ditampilkan dengan dua isosurfaces: permukaan buram berdensitas tinggi dan permukaan transparan berdensitas rendah. Sebagai alternatif, distribusi divisualisasikan dengan DVR dan kode warna. Untuk jenis visualisasi ini, bidang potong digunakan untuk mengungkap inti distribusi (Gambar 9c). Karena molekul bukanlah benda kaku, Skånberg dkk. memperkenalkan plot variasi bentuk untuk menilai apakah asumsi benda kaku mendekati benar. Ruang bentuk barisentris plot tersebut mencakup tiga dimensi: linier, planar, dan bulat. Dalam plot bentuk ini, molekul kaku hanya mencakup area kecil, sedangkan molekul fleksibel mencakup area yang lebih luas. Metode ini berlaku untuk aplikasi yang lebih luas, dan struktur referensi tidak terbatas pada jenis molekul tertentu, misalnya, struktur referensi dalam makalah tersebut mencakup protein, DNA, dan molekul kecil. Dalam VIAMD [ SHYL23 ], visualisasi ini tertanam dalam alat analisis visual yang lebih komprehensif untuk MD.
Representasi alternatif ketidakpastian dipilih dalam aplikasi analitik visual InVADo oleh Sch äfer et al. [ SBB*24 ], yang memungkinkan data docking dianalisis dan dieksplorasi. Untuk menyelidiki probabilitas docking di situs pengikatan, molekul ligan yang di-dock pertama-tama dikelompokkan. Bagian permukaan molekul yang dekat dengan salah satu posisi klaster ini disorot oleh warna dan opasitas untuk menunjukkan kemungkinan situs pengikatan. Jumlah kemungkinan interaksi juga dapat dipetakan warna ke permukaan protein untuk menunjukkan probabilitas spasial suatu interaksi. Ini dapat dilakukan untuk berbagai jenis interaksi (yaitu, sifat fisika-kimia). Distribusi ikatan hidrogen yang mungkin ditunjukkan sebagai glif kerucut yang terhubung ke atom protein yang berpartisipasi. InVADo juga menawarkan visualisasi non-spasial untuk menunjukkan ketidakpastian hasil docking: Distribusi skor docking (yang menunjukkan probabilitas kejadian docking yang sesuai) per klaster ditunjukkan sebagai kotak plot dan berdasarkan warna dalam peta panas tersegmentasi. Frekuensi interaksi yang berbeda dalam suatu klaster ditampilkan sebagai diagram batang untuk memungkinkan pengguna menganalisis interaksi yang paling mungkin.
8. Asam NukleatGambar
Sebagian besar makalah tentang asam nukleat berfokus pada visualisasi RNA. Hanya dua makalah yang secara khusus memvisualisasikan DNA.
8.1. Asam amino
Semua makalah tentang RNA mengodekan ketidakpastian struktur sekunder Gambardengan menggunakan warna Gambar. Struktur sekunder molekul RNA—khususnya, penyelarasan basa—biasanya divisualisasikan dengan hanya menampilkan penyelarasan yang paling mungkin. Basa-basa direpresentasikan dengan huruf-huruf yang sesuai dan disusun sedemikian rupa sehingga basa-basa yang berpasangan diposisikan berdekatan satu sama lain. Selain itu, basa-basa yang berpasangan sering dihubungkan dengan garis untuk menunjukkan interaksinya. ViennaRNA [ GLB*08 ]—perangkat untuk prediksi dan analisis struktur sekunder RNA—menggabungkan UV dasar dengan menyediakan opsi untuk mewarnai basa-basa menurut probabilitas pasangan basanya (Gambar 3a ). CentroidFold [ SHAM09 ] adalah server web yang menawarkan opsi visualisasi serupa. Di luar visualisasi ketidakpastian sederhana ini, yang mendukung tugas-tugas ikhtisar dan analisis dasar, ada juga metode berdasarkan diagram pelangi yang memfasilitasi tugas-tugas perbandingan. Diagram pelangi terdiri dari urutan pasangan basa yang ditulis dalam satu baris dengan busur yang menghubungkan basa-basa yang berpasangan. RNABows [ AJ13 ] menyempurnakan diagram pelangi dengan informasi ketidakpastian dengan mengodekan probabilitas pasangan basa dalam lebar dan kecerahan pita. Pasangan yang tidak mungkin dihubungkan dengan garis yang lebih tipis dan lebih terang dan dengan demikian kurang terlihat daripada pasangan yang mungkin. Selain itu, RNABows menawarkan kemungkinan untuk membandingkan dua sekuens dengan memplotnya kembali ke belakang. Léger et al. [ LCT19 ] mengadaptasi RNABows dengan menggunakan lingkaran sebagai dasar untuk memplot sekuens (lihat Gambar 3b ). Beberapa sekuens dibandingkan dengan memplot lingkaran masing-masing di samping satu sama lain. Ini memungkinkan perbandingan lebih dari dua sekuens, tetapi hubungan di antara keduanya tidak sejelas dalam RNABows [ AJ13 ]. Irving dan Weeks [ IW24 ] mengusulkan alat RNAvigate untuk membandingkan prediksi pasangan basa dari beberapa penelitian. Mereka mengadaptasi plot pelangi dan mewarnai pita sesuai dengan jumlah penelitian yang memprediksi pasangan tertentu. Selain itu, pasangan energi bebas minimum (MFE) ditunjukkan di bawah sekuens dengan warna abu-abu.
8.2.Analisis DNA
Ertl et al. [ EKK*14 ] memvisualisasikan kerapatan ion di dekat untai DNA dalam nanopore menggunakan permukaan semitransparan bersarang. Karena DNA bermuatan negatif, ion positif cenderung berada di dekat DNA (lihat Gambar 11 ). Artinya, isosurfaces yang berbeda menggambarkan rentang kerapatan atau probabilitas ion yang berbeda. Warna berurutan yang berbeda (biru, hijau, kuning) diberikan untuk memudahkan pembedaan permukaan bersarang. Selain itu, pengguna dapat memvisualisasikan garis lintasan ion. Xie et al. [ XZF20 ] mengusulkan teknik untuk visualisasi DNA yang sadar ketidakpastian. Alat mereka PyShifts memfasilitasi perbandingan antara pergeseran kimia yang diukur secara eksperimental dan yang dihitung. Pergeseran ini memberikan informasi tentang keadaan konformasi biomolekul dan dapat digunakan untuk memodelkan struktur sekunder dan tersiernya. Besarnya perbedaan dalam pergeseran yang diukur dan dihitung dikodekan dalam ukuran bola yang digambar pada posisi nukleus. Warna bola menunjukkan tanda perbedaan. Metode untuk memvisualisasikan DNA lebih generik daripada metode RNA yang diusulkan dan juga dapat diterapkan pada jenis biomolekul lainnya.
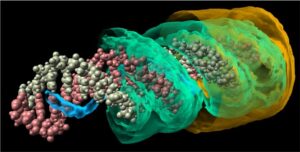
Berikut ini, kita akan membahas pola dan tren teknik UV untuk struktur biomolekuler. Gambar 12 menunjukkan plot set paralel [ KBH06 ] dari distribusi dan korelasi atribut literatur yang dikumpulkan sepanjang dimensi utama skema kita. Dalam visualisasi, dimensi—jenis molekul, manifestasi, dan pemetaan—diwakili dengan kontribusi total yang konsisten di seluruh lapisan. Namun, makalah sering kali bervariasi dalam jumlah kategori yang berlaku per dimensi. Kami mendistribusikan kontribusi setiap makalah secara merata di antara kategori yang berlaku dalam dimensi tertentu. Misalnya, jika sebuah makalah menggunakan tiga strategi pemetaan, setiap pemetaan menerima lebar proporsional 1/3 dari total kontribusi makalah. Sebaliknya, makalah dengan hanya satu pemetaan mengalokasikan kontribusi penuhnya ke pemetaan itu. Strategi ini memastikan bahwa setiap makalah berkontribusi secara merata pada total lebar per dimensi.
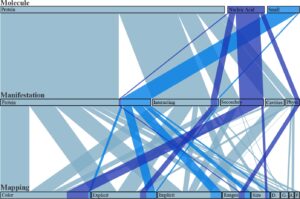
9.1. Jenis Molekul
Bidang visualisasi struktur biomolekuler yang sadar ketidakpastian dengan jelas menekankan protein, dengan sebagian besar makalah yang diterbitkan berfokus pada jenis biomolekul ini. Ini bukan masalah utama untuk molekul kecil Gambar, karena sebagian besar metode alternatif mudah diterapkan. Molekul kecil tidak memiliki manifestasi ketidakpastian yang unik, memastikan bahwa pendekatan yang ada umumnya berlaku tanpa adaptasi yang signifikan. Namun, representasi asam nukleat Gambarjuga terbatas, terutama untuk DNA. Ini adalah masalah yang lebih signifikan karena asam nukleat memiliki kebutuhan visualisasi yang unik—misalnya, untuk probabilitas pasangan basa—yang memerlukan pengembangan visualisasi ketidakpastian tertentu. Menariknya, kecuali untuk makalah oleh Ertl et al. [ EKK*14 ] tentang DNA, dan kerangka kerja yang lebih umum yang berlaku untuk berbagai macam molekul yang diusulkan oleh Skanberg et al. [ SFL*21 , SHYL23 ], sebagian besar makalah tentang asam nukleat berasal dari pakar domain dalam bioinformatika dan bidang terkait, bukan dari komunitas penelitian visualisasi. Karbohidrat dan lipid juga kurang terwakili. Ada beberapa karya mengenai karbohidrat [ BGB*18 , BWR*20 , BGB*20 ] dan lipid [ DCS20 , ST21 ]. Akan tetapi, karya-karya tersebut terutama berkaitan dengan interaksi protein dan dengan demikian diurutkan ke dalam kategori protein skema kita. Fokus ini agak diharapkan, karena visualisasi karbohidrat atau lipid individual cenderung memiliki minat yang terbatas. Sebaliknya, relevansi utama terletak pada interaksinya dengan molekul lain, seperti protein, atau, dalam kasus lipid, perilaku kolektifnya dalam rakitan molekul yang lebih besar seperti membran.
Fokus pada protein Gambarselaras dengan penekanan dalam ilmu domain. Secara historis, dan pada tingkat yang lebih rendah bahkan saat ini, protein telah menjadi fokus utama penelitian [ Cam02 , BBD*22 ]. Fokus ini dapat dikaitkan dengan beberapa faktor, seperti ketersediaan data struktural. Protein, misalnya, umumnya lebih mudah dikristalkan daripada RNA karena RNA lebih fleksibel [ MSAC15 ]. Selain itu, protein memainkan peran sentral sebagai molekul fungsional dalam sel [ AHJ*22 ]. Namun, fokus ini secara bertahap bergeser seiring waktu, terutama karena RNA tidak lagi dipandang terutama sebagai molekul pembawa pesan [ MM14 ]. Selain itu, kemajuan dalam teknik seperti cryo-EM telah memungkinkan visualisasi sistem molekuler yang lebih besar, seperti membran. Ini telah memperluas cakupan penelitian di luar molekul dan protein kecil yang relevan secara farmasi untuk mencakup rentang biomolekul yang lebih luas dan skala struktural yang lebih besar, seperti seluruh membran atau kompleks molekuler besar [ RCC*18 ]. Namun, pendekatan yang sadar ketidakpastian untuk visualisasi molekuler skala besar (misalnya mesoskala) dan multiskala sebagian besar masih belum dieksplorasi. Beberapa kemajuan telah dibuat di area terkait, seperti dinamika sistem membran. Misalnya, Chavent et al. [ CRG*14 ] menggunakan pathlines, arrow glyphs, dan streamlines untuk memvisualisasikan aliran lipid dalam sistem membran. Seiring dengan kemajuan biologi struktural dalam beberapa dekade terakhir, komunitas penelitian visualisasi perlu mengatasi meningkatnya kompleksitas kumpulan data yang lebih besar dan sistem molekuler yang lebih rumit.
9.2. Manifestasi Ketidakpastian
Ketidakpastian dalam visualisasi molekuler muncul dalam beberapa bentuk, tergantung pada molekul atau sistem yang dipelajari. Fokus utama adalah pada ketidakpastian posisi Gambar. Seperti yang dibahas dalam Bagian 4.2, informasi ketidakpastian posisi tersedia dengan mudah dan biasanya menjadi salah satu manifestasi pertama ketidakpastian yang dipertimbangkan. Tidak mengherankan, mayoritas metode visualisasi berfokus pada ketidakpastian posisi. Selain itu, metode yang dikembangkan untuk satu jenis molekul sering kali dapat ditransfer ke yang lain saat menyelidiki ketidakpastian posisi. Lebih jauh lagi, ketidakpastian posisi memengaruhi sebagian besar sifat lain yang diselidiki untuk biomolekul dan menyebar, yang memengaruhi ukuran ketidakpastian untuk manifestasi lain juga. Sifat fisiko-kimia Gambaradalah area minat lainnya, dengan visualisasi biasanya menggunakan metode generik seperti representasi distribusi langsung. Meskipun teknik-teknik ini berlaku secara luas, teknik-teknik ini tidak disesuaikan dengan sifat-sifat tertentu, yang dapat membatasi efektivitasnya dalam beberapa konteks. Interaksi Gambardengan molekul lain adalah topik penting tetapi kurang dieksplorasi dalam UV. Jika interaksi ditangani, pekerjaan yang ada terutama difokuskan pada protein dan terutama membahas interaksi dengan molekul-molekul kecil di sekitarnya, seperti air. Akan tetapi, banyak jenis interaksi lain memerlukan visualisasi ketidakpastian yang sesuai, termasuk tetapi tidak terbatas pada protein-protein, protein-ligan, asam nukleat-protein, ligan-asam nukleat, dan interaksi membran. Kemajuan sedang dibuat dalam memvisualisasikan jenis interaksi lain ini, seperti yang ditunjukkan oleh alat analisis visual yang dikembangkan oleh Furmanová et al. [ FJK*20 ] untuk ansambel besar kompleks protein multitubuh. Akan tetapi, topik ini sebagian besar masih kurang dieksplorasi. Seiring dengan peningkatan daya komputasi dan peningkatan teknik pencitraan, penelitian telah bergeser ke arah mempelajari sistem molekuler yang lebih dinamis dan kompleks, yang menyoroti perlunya pengembangan lebih lanjut di area ini. Untuk ketidakpastian struktur sekunder Gambar, upaya visualisasi untuk RNA terutama difokuskan pada probabilitas pasangan basa. Visualisasi ini cukup mendasar—seperti pemetaan warna yang diterapkan pada representasi struktur 2D yang ada—atau terbatas pada visualisasi urutan linier. Visualisasi 3D yang komprehensif untuk ketidakpastian struktur sekunder RNA sama sekali tidak ada. Selain itu, visualisasi ruang konformasi tidak ada, yang menyoroti kesenjangan yang signifikan dalam menangkap cakupan penuh variabilitas struktural. UAV untuk rongga biomolekulerGambardalam protein juga kurang mendapat perhatian. Sebagian dari hal ini kemungkinan disebabkan oleh rongga yang merupakan manifestasi ketidakpastian yang relatif terspesialisasi. Alat yang ada untuk memvisualisasikan rongga sering kali mengandalkan representasi 2D yang agak abstrak untuk ketidakpastian. Hal ini tidak mengejutkan, karena rongga pada dasarnya kompleks karena ketergantungannya pada ketidakpastian posisi dan sifat fisikokimia. Dengan demikian, sangat penting untuk mengevaluasi metode yang ada dengan benar untuk menentukan apakah metode tersebut berhasil membantu membuat fenomena kompleks ini dapat dipahami dan apakah pemetaan abstrak yang diusulkan sesuai.
9.3. Pemetaan
Dalam memvisualisasikan ketidakpastian struktur molekul, pemetaan distribusi eksplisit dan implisit, bersama dengan penggunaan atribut seperti warna, merupakan teknik yang paling umum digunakan. Visualisasi ensemble Gambarsangat populer, yang tidak mengherankan mengingat banyak metode pencitraan dan simulasi menghasilkan serangkaian konformasi. Hal ini menjadikan visualisasi ensemble sebagai pilihan alami untuk merepresentasikan ketidakpastian. Demikian pula, Gambarvisualisasi distribusi eksplisit sering kali secara langsung menggambarkan peta kerapatan elektron atau distribusi sifat fisikokimia, yang menyediakan pendekatan langsung lainnya. Selain itu, Gambarpemetaan warna sangat umum karena sederhana, diterapkan dalam perangkat lunak visualisasi standar, dan mudah diterapkan.
Meskipun pendekatan-pendekatan ini sudah mapan, teknik-teknik lain masih relatif belum dieksplorasi. Misalnya, animasi Gambartidak digunakan untuk memvisualisasikan ketidakpastian dalam biomolekul, kecuali untuk salah satu makalah paling awal tentang subjek tersebut [ FTPG90 ]. Salah satu kemungkinan alasannya adalah bahwa biomolekul pada dasarnya adalah struktur yang dinamis, dan animasi biasanya diharapkan untuk menggambarkan gerakan molekuler daripada ketidakpastian. Sementara animasi yang menggabungkan gerakan halus, seperti goyangan, terkadang digunakan dalam komunikasi sains molekuler, penyelidikan yang tepat dan sistematis tentang kemungkinan penggunaan animasi untuk memvisualisasikan ketidakpastian masih kurang. Strategi pemetaan lain yang belum dieksplorasi adalah glif Gambar. Sementara Bryden et al. [ BPG12 ] memperkenalkan pendekatan berbasis glif, teknik-teknik ini belum diadopsi secara luas. Hal ini menyisakan potensi yang cukup besar bagi glif untuk digunakan, terutama dalam visualisasi statis atau untuk komunikasi sains. Demikian pula, representasi statis dinamika lainnya seperti garis lintasan Gambar, garis gerak, atau gerakan kabur jarang digunakan dan sebagian besar masih belum dieksplorasi untuk menggambarkan ketidakpastian dalam sistem molekuler.
Contoh motion blur untuk visualisasi dinamika molekuler dapat ditemukan dalam makalah oleh Eschner et al. [ EMW23 ], yang menggunakan motion blur untuk mengarahkan perhatian dalam visualisasi dinamika molekuler. Spalvieri et al. [ SML*22 ] mengilustrasikan gerakan molekuler menggunakan motion blur untuk untai DNA dalam makalah mereka tentang kolaborasi dengan desainer untuk meningkatkan visualisasi molekuler. Contoh hasil lain dari kolaborasi tersebut adalah visualisasi temporal protein membran yang berfungsi sebagai saluran ion melalui penghalang membran; visualisasi 2D semi-transparan yang abstrak dari penyempitan utama saluran ditumpangkan untuk membentuk gambar 2D tunggal, mirip dengan pendekatan garis kontur Byška et al. [ BJG*15 ]. Jenis kolaborasi ini dapat menginspirasi ide-ide baru untuk UV molekuler. Baru-baru ini, penelitian telah mengeksplorasi atribut visual alternatif untuk pemetaan ketidakpastian, seperti kepadatan Gambardalam tekstur ilustrasi [ SMCL24 ] dan atribut garis yang berbeda [ SLK*23 ]. Metode-metode ini dapat menampilkan statistik ringkasan sambil membiarkan saluran warna terbuka untuk properti lainnya. Namun, kami percaya masih ada potensi signifikan untuk eksplorasi lebih lanjut terhadap atribut alternatif, terutama melalui kolaborasi dengan para ahli desain.
Kami mengklasifikasikan strategi pemetaan ke dalam dua kategori utama: representasi visual dari distribusi ketidakpastian dan representasi visual dari statistik ringkasan. Visualisasi distribusi khususnya berguna untuk memahami ketidakpastian yang melekat pada biomolekul, yang sering kali diterjemahkan menjadi pemahaman ruang konformasi potensial yang dapat ditempati oleh molekul tertentu. Statistik ringkasan biasanya dapat dengan mudah diintegrasikan ke dalam visualisasi yang ada, menawarkan informasi ketidakpastian dengan cara yang tidak terlalu mengganggu sambil hanya menyediakan data agregat. Namun, visualisasi distribusi yang tersedia saat ini sering kali sangat berantakan dan sulit dipahami.
Upaya di area terkait dapat menjadi inspirasi untuk teknik yang lebih maju. Liu et al. [ LLBP12 ], misalnya, menggunakan model campuran Gaussian untuk merepresentasikan distribusi data per voksel dalam suatu volume. Rendering probabilistik kemudian dapat digunakan untuk memvisualisasikan ketidakpastian. Untuk rendering dinamis, volume ditampilkan dengan mengambil sampel distribusi pada setiap bingkai, di mana area dengan ketidakpastian yang lebih tinggi muncul sebagai peningkatan kedipan dibandingkan dengan area dengan ketidakpastian yang lebih rendah. Untuk gambar statis, sampel dapat diintegrasikan dalam ruang layar, menghasilkan lebih banyak keburaman di area dengan ketidakpastian yang lebih tinggi.
Area lain yang kurang mendapat perhatian adalah perbandingan molekul serupa yang sadar ketidakpastian. Perbandingan struktur serupa—seperti tipe liar dan struktur bermutasi—sangat relevan. Aplikasi potensial lain untuk visualisasi komparatif tersebut adalah ensembel lintasan MD, yang diidentifikasi Belghit et al. [ BSD*24 ] sebagai salah satu tantangan utama untuk visualisasi MD. Meskipun demikian, UV untuk perbandingan tersebut masih langka. Dalam karya yang ada, beberapa struktur ditampilkan melalui overlay langsung—yaitu, sebagai ensembel—atau sebagai visualisasi berdampingan. Kocincová et al. [ KJB*17 ] mengusulkan pendekatan yang lebih maju dengan menggabungkan tampilan ensembel khas dengan representasi 1D yang disandingkan atau ditumpangkan dari struktur sekunder protein.
Pemetaan ketidakpastian dalam biomolekul dapat memperoleh manfaat dari teknik visualisasi yang lebih abstrak atau ilustratif, suatu area yang sebagian besar masih belum dimanfaatkan. Sementara pendekatan abstraksi tidak memungkinkan penilaian yang tepat terhadap tingkat ketidakpastian, pendekatan tersebut dapat mewakili kategori ketidakpastian yang luas, seperti yang diilustrasikan oleh Strothotte et al. [ SMI99 ] untuk rekonstruksi arsitektur kuno. Sepengetahuan kami, Sorger et al. [ SMK*16 ] adalah satu-satunya penulis yang telah menyajikan makalah menggunakan abstraksi dalam konteks terkait ketidakpastian untuk biomolekul. Mereka melakukan interpolasi antara berbagai keadaan model mesoskala biologis pada berbagai tingkat detail dengan menerapkan teknik abstraksi. Model dengan tingkat detail yang lebih tinggi diabstraksikan hingga memiliki resolusi yang mirip dengan model yang kurang detail. Pada tingkat detail ini, model dapat diinterpolasi dan kemudian ditransformasikan balik ke tingkat abstraksi asli. Jadi, meskipun pendekatan ini tidak menggunakan abstraksi untuk menyorot wilayah dengan ketidakpastian yang lebih tinggi dalam gambar yang sama, konsep tersebut juga dapat diterapkan pada UV. Pendekatan tambahan untuk menciptakan visualisasi abstrak atau ilustratif biomolekul ada untuk tujuan lain. Parulek dkk. [ PJR*14 ] menggunakan abstraksi visual yang mulus untuk meningkatkan kinerja rendering. Van der Zwan dkk. [ vLBI11 ] menggunakan abstraksi ilustratif berkelanjutan untuk fokus dan konteks serta untuk mengarahkan perhatian. Jadi, meskipun metode untuk ilustrasi dan abstraksi ada, kegunaannya untuk UV masih belum dieksplorasi.
9.4. Ketersediaan Perangkat Lunak
Secara umum, ketersediaan perangkat lunak untuk visualisasi struktur biomolekuler yang sadar ketidakpastian buruk. Hanya untuk setengah dari proyek (30 dari 61), perangkat lunak disediakan untuk mereproduksi visualisasi. Hanya 25 proyek yang masih tersedia. Ini menghadirkan masalah yang signifikan, karena pentingnya UV sering ditekankan dalam literatur [ Joh04 , BHJ*14 , PKH21 ]. Namun, pengembangan metode ini saja tidak cukup untuk adopsi mereka. Untuk memastikan kegunaan, praktisi harus memiliki akses ke alat-alat ini. Idealnya, pendekatan baru harus diintegrasikan ke dalam kerangka kerja visualisasi yang digunakan secara luas seperti PyMOL [ Sch ], VMD [ HDS96 ], dan ChimeraX [ PGH*21 ]. Minimal, kode sumber yang dapat direproduksi harus disediakan untuk mendukung penggunaan dan validasi yang lebih luas.
Namun, beberapa pemetaan tersedia dalam perender molekuler standar dan digunakan secara luas. Pemetaan warna nilai ketidakpastian skalar, seperti faktor-b, umumnya dapat diakses dan sering diterapkan. Visualisasi ensembel sederhana juga umum. Metafora sosis, yang memvariasikan radius tabung tulang punggung berdasarkan ketidakpastian, tersedia di banyak perender. DVR dan ekstraksi isolevel dari distribusi probabilitas juga didukung secara luas. Selain itu, beberapa perender menyertakan opsi untuk menampilkan glif, seperti vektor gerakan untuk NMA. Namun, ketersediaan metode atau solusi yang lebih canggih untuk manifestasi ketidakpastian yang kurang umum sering kali buruk.
9.5. Evaluasi
Kesenjangan yang signifikan adalah evaluasi metode yang diusulkan. Sebagian besar metode dinilai terutama melalui studi kasus. Jika penulis bukan pakar domain, wawancara pakar sering kali melengkapi evaluasi. Meskipun studi kasus berharga untuk menunjukkan bahwa suatu metode dapat menawarkan keuntungan dibandingkan teknik visualisasi tradisional, studi kasus jarang mencukupi untuk evaluasi yang komprehensif. Hanya lima metode yang menjadi sasaran evaluasi kuantitatif yang lebih ketat, seperti studi pengguna atau studi persepsi. Kurangnya evaluasi empiris ini mempersulit pengambilan keputusan yang tepat tentang metode UV. Pemahaman yang lebih mendalam tentang kekuatan dan kelemahan berbagai pendekatan umumnya diperlukan untuk memilih metode yang tepat untuk masalah tertentu.
9.6. Teknik Lintas Domain
Taksonomi untuk UV sering mengklasifikasikan metode berdasarkan dimensionalitas data [ PWL97 , BAOL12 , BHJ*14 ]. Sebagian besar metode yang dibahas dalam laporan tentang UV umum berfokus pada data 1D atau 2D. Data struktur biomolekuler secara inheren 3D. Dalam visualisasi data 1D atau 2D yang tidak pasti, ketidakpastian sering kali direpresentasikan dengan menggabungkan dimensi tambahan. Namun, pendekatan ini biasanya tidak praktis untuk 3D, karena semua dimensi spasial yang tersedia sudah digunakan. Oleh karena itu, pemetaan ketidakpastian alternatif harus digunakan. Karena itu, banyak metafora lain yang digunakan untuk ketidakpastian dalam data berdimensi lebih rendah dapat diadaptasi untuk data 3D. Misalnya, batang kesalahan untuk estimasi titik dalam 1D menjadi pita kesalahan dalam 2D dan lambung dalam 3D. Kemampuan beradaptasi ini berarti metafora dari taksonomi umum sering kali dapat menginformasikan UV struktur biomolekuler. Beberapa metafora yang diusulkan dalam literatur, meskipun belum diterapkan pada struktur biomolekuler, juga dapat dieksplorasi untuk data struktur biomolekuler. Contohnya termasuk kedipan atau osilasi [ PWL97 , BHJ*14 ], sonifikasi [ PWL97 , BAOL12 ], dan perpindahan permukaan atau benjolan [ PWL97 , BHJ*14 ]. Misalnya, Grigoryan dan Rheingans [ GR04 ] membuat permukaan berisik dengan merepresentasikan permukaan sebagai kumpulan titik yang dipindahkan berdasarkan ketidakpastian struktur. Teknik ini dapat langsung diadaptasi untuk permukaan molekuler. Dengan demikian, visualisasi struktur biomolekuler dapat memperoleh manfaat dari teknik dan wawasan dari disiplin ilmu lain. Sebaliknya, kebalikannya juga berlaku: Banyak metode yang dikembangkan untuk visualisasi struktur biomolekuler yang sadar ketidakpastian tidak terbatas pada domain ini dan dapat diterapkan ke bidang terkait, seperti ilmu material, teknik kimia, dan nanoteknologi. Lebih jauh lagi, disiplin ilmu yang mengandalkan model 3D dengan ketidakpastian yang melekat, seperti pencitraan medis, menghadapi tantangan serupa dan dapat memperoleh manfaat dari pendekatan bersama. Data ensemble, salah satu bentuk data struktur biomolekuler tak tentu yang paling umum, merupakan bagian penting dari banyak bidang lain seperti meteorologi, klimatologi, astrofisika, fisika energi tinggi, dan oseanografi [ WHLS19 ], yang menunjukkan peluang untuk kolaborasi lintas disiplin dan transfer metode. UV untuk struktur biomolekuler merupakan bidang penelitian yang aktif, didukung oleh ketersediaan data yang luas dan pentingnya data tersebut bagi ilmu hayati. Pengembangan teknik visualisasi untuk domain ini meningkatkan interpretasi data biomolekuler dan memberikan wawasan yang berlaku untuk tantangan UV yang lebih umum, termasuk visualisasi ensemble dan data 3D tak tentu.
10. Arah Penelitian
Pada bab sebelumnya, kami telah menjajaki tren dan menguraikan berbagai tantangan di bidang ini. Di sini, kami akan mempersempit fokus dan menyoroti beberapa area yang kami anggap paling penting untuk penelitian di masa mendatang.
Sistem Kompleks dan Multiskala Kemajuan dalam teknologi pencitraan memungkinkan visualisasi sistem yang semakin kompleks. Sementara beberapa pendekatan ada untuk memvisualisasikan sistem molekul yang berinteraksi, seperti interaksi protein-ligan, masih ada ruang yang signifikan untuk perbaikan. UAV untuk sistem yang lebih besar, seperti struktur mesoskala atau visualisasi yang mencakup beberapa skala, sebagian besar masih belum dieksplorasi. Metode yang bekerja dengan baik untuk struktur molekul berukuran kecil hingga sedang sering menjadi berantakan atau tidak praktis ketika diterapkan pada sistem yang lebih kompleks. Ini menyoroti perlunya pendekatan yang dapat secara efektif mengelola skala dan kompleksitas data ini. Solusi potensial adalah mengadopsi teknik visualisasi yang lebih abstrak dan ilustratif. Pendekatan ini dapat menawarkan visualisasi yang lebih jelas dan lebih dapat ditafsirkan sambil mengatasi tantangan unik dari sistem molekuler yang kompleks.
Ruang Konformasional Meskipun sudah ada sejumlah besar literatur yang memvisualisasikan ruang konformasional, masalah ini tetap menantang, terutama untuk molekul yang lebih besar, karena teknik yang ada sering kali menjadi berantakan dan sulit ditafsirkan. Upaya awal untuk meningkatkan visualisasi ini dilakukan oleh Melvin et al. [ MS16 ], yang menggunakan fokus dan konteks, serta subset konformasi yang dipilih secara sengaja, untuk mengurangi kompleksitas visual. Meskipun demikian, visualisasi yang mudah dipahami dari seluruh ruang konformasi masih langka. Mirip dengan visualisasi sistem yang kompleks, volume dan kompleksitas data dapat sangat banyak. Namun, sifat multidimensi ruang konformasional menghadirkan tantangan tambahan. Seperti halnya sistem yang kompleks, teknik abstraksi dan ilustrasi menawarkan solusi yang menjanjikan. Representasi yang disederhanakan yang menekankan status atau transisi utama dapat meningkatkan kejelasan, sementara alat interaktif untuk menjelajahi wilayah tertentu dari lanskap konformasi dapat meningkatkan kegunaan. Sebagai alternatif, animasi—mirip dengan HOP [ Hul16 ]—dapat secara dinamis mewakili perubahan konformasi, mengurangi kebutuhan untuk menyampaikan semua informasi dalam satu visualisasi statis.
Teknik Perbandingan Membandingkan konfigurasi molekuler dan ketidakpastiannya biasanya melibatkan visualisasi berdampingan atau overlay sederhana. Metode ini efektif untuk set data kecil tetapi menjadi tidak praktis saat jumlah molekul meningkat. Overlay menghadapi tantangan yang mirip dengan memvisualisasikan ruang konformasi, seperti menjadi berantakan dan sulit ditafsirkan. Masalah ini terutama bermasalah saat membandingkan molekul dinamis. Ensemble simulasi dinamika molekuler (MD) telah menjadi penting untuk mengatasi kurangnya reproduktifitas dalam simulasi individual [ WSC21 ]. Namun, ensembel MD menghasilkan sejumlah besar data, yang membutuhkan teknik visualisasi baru yang mampu menangani kompleksitas ini tanpa membebani pengguna. Tantangan ini juga disoroti oleh Belghit et al. [ BSD*24 ]. Masalah skalabilitas ini tidak terbatas pada sistem biomolekuler; ini juga memengaruhi metode visualisasi ensembel yang lebih luas [ WHLS19 ]. Kemajuan di kedua bidang dapat menawarkan manfaat bersama, yang mengarah pada solusi yang lebih baik untuk memvisualisasikan set data yang besar dan kompleks.
Asam Nukleat Gambar Meskipun menjadi fokus dari banyak topik penelitian saat ini [ WEFC21 , BJZ*22 , WD23 ], asam nukleat sering kurang terwakili dalam hal teknik UV khusus. Visualisasi saat ini terutama berfokus pada probabilitas pasangan basa dan bergantung pada metode yang relatif sederhana. Sementara pendekatan ini memberikan beberapa wawasan tentang struktur asam nukleat, cakupannya terbatas dan gagal menangkap kompleksitas penuh dari molekul-molekul ini. Visualisasi 3D yang komprehensif, yang dapat memberikan representasi yang lebih lengkap dari struktur asam nukleat, sebagian besar tidak ada. Selain itu, memvisualisasikan ruang konformasi asam nukleat masih merupakan kesenjangan yang signifikan meskipun penting untuk memahami dinamika molekuler, interaksi, dan fungsi.
Evaluasi Teknik UV untuk struktur biomolekuler tidak dievaluasi dengan cukup ketat. Misalnya, sangat penting untuk menentukan metode mana yang paling intuitif bagi pemirsa, seberapa efektif mereka mendukung inferensi nilai kuantitatif, dan apakah mereka secara akurat mewakili ruang konformasi biomolekul. Karena manusia sering bernalar dengan cara yang tidak intuitif ketika menafsirkan ketidakpastian [ PKH21 ], sangat penting untuk mengevaluasi visualisasi ketidakpastian secara ketat untuk memastikan mereka secara efektif mendukung pemahaman dan pengambilan keputusan yang akurat. Mengevaluasi metode UV secara inheren menantang [ Hul16 ], bahkan untuk visualisasi 1D dan 2D yang sederhana. Menggabungkan dan mengevaluasi ketidakpastian dalam konteks visualisasi struktur biomolekuler 3D bahkan lebih kompleks. Namun, mengatasi kesenjangan ini sangat penting. Ini akan memungkinkan praktisi untuk memilih metode yang paling sesuai untuk masalah tertentu, mengidentifikasi kekurangan dalam teknik yang ada, dan mendorong pengembangan metode baru yang lebih memenuhi tuntutan UAV.
11. Kesimpulan
Dalam laporan ini, kami menyajikan pendekatan UAV untuk struktur biomolekuler, mengklasifikasikan metode berdasarkan tiga dimensi utama: jenis molekul, manifestasi ketidakpastian, dan strategi pemetaan. Meskipun metode ini menunjukkan strategi untuk merepresentasikan ketidakpastian untuk molekul individual, metode ini sering kali gagal memenuhi tuntutan penelitian biomolekuler modern. Ukuran dan kompleksitas kumpulan data biomolekuler yang terus meningkat memerlukan teknik visualisasi yang dapat diskalakan secara efektif dan tetap dapat diinterpretasikan. Pendekatan saat ini sering kali tampak berantakan bahkan untuk molekul berukuran kecil hingga sedang, sehingga tidak cocok untuk sistem yang lebih besar seperti data mesoskala atau kumpulan struktur dinamis. Kesenjangan ini menyoroti perlunya strategi visualisasi inovatif yang dapat menangani kompleksitas dan skala data biomolekuler modern. Memajukan metode UAV sangat penting untuk meningkatkan cara peneliti menginterpretasikan dan berinteraksi dengan data biomolekuler yang kompleks, memungkinkan wawasan yang lebih jelas dan pengambilan keputusan yang lebih tepat. Mengingat adanya kesamaan dengan domain lain, seperti ketidakpastian umum dan visualisasi kumpulan, kemajuan dalam UAV untuk struktur biomolekuler kemungkinan akan memiliki dampak yang lebih luas, yang menguntungkan seluruh bidang UAV.
